Credito:Wiley
Che si tratti di rivelare un perpetratore con prove del DNA, diagnosticare un agente patogeno, classificare una scoperta paleontologica, o determinare la paternità, la duplicazione degli acidi nucleici (amplificazione) è indispensabile. Nel diario Angewandte Chemie , gli scienziati hanno ora introdotto un nuovo, molto semplice, ma metodo altamente sensibile e affidabile che evita le normali fasi di riscaldamento e raffreddamento, così come strumenti complicati. I reagenti possono essere liofilizzati, consentendo di utilizzare questo metodo universale al di fuori del laboratorio.
Il metodo di amplificazione più comunemente usato è la reazione a catena della polimerasi (PCR), che si basa sulla ripetizione di più cicli termici in appositi strumenti che hanno un'elevata richiesta di potenza. È difficile eseguire al di fuori di un laboratorio, al capezzale di un paziente o in un luogo remoto, Per esempio. I metodi alternativi senza cicli termici sono spesso complicati o non sufficientemente sensibili, richiedono reagenti costosi, o non sono ampiamente applicabili.
I ricercatori che lavorano con Bin-Cheng Yin e Bang-Ce Ye presso la East China University of Science &Technology, Shanghai, Cina, ora hanno sviluppato un nuovo, metodo economico:chiamato reazione di amplificazione basata su Cas9n (Cas9nAR), consiste in un unico passaggio in soluzione omogenea, e avviene ad una temperatura costante di 37 °C.
In questo approccio i ricercatori utilizzano componenti del "sistema immunitario" dei batteri. Quando i batteri vengono infettati da un virus, Per esempio, tagliano il materiale genetico estraneo in piccoli pezzi e lo introducono in aree specifiche del proprio genoma. In caso di successiva infezione, i filamenti di RNA del batterio "riconosceno" queste sequenze e dirigono speciali "forbici genetiche" per tagliare il DNA estraneo. Questi strumenti sono stati impiegati anche nella moderna ingegneria genetica.
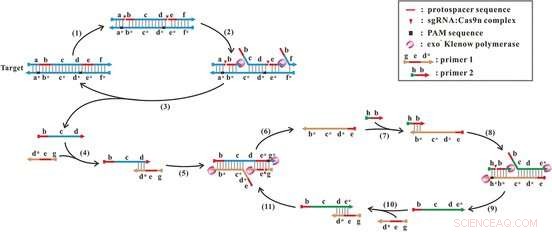
Yin, Voi, e i loro collaboratori hanno alterato la forbice genetica nota come Cas9 in modo che non tagli più completamente il DNA. Anziché, taglia un solo filo, introducendo un "nick". Questo tipo di enzima è chiamato "nickasi". Come nel sistema batterico, La nickasi Cas9 si lega a un filamento di RNA, che determina la posizione del nick. Questo RNA può essere fatto in modo che riconosca una sequenza di DNA caratteristica di un agente patogeno, Per esempio. La nickasi Cas9 poi intacca il DNA immediatamente adiacente.
Per la nuova tecnica, i ricercatori hanno prodotto due diversi complessi di RNA della nickasi Cas9, che intaccano il DNA in due punti diversi. Una polimerasi comunemente usata nella PCR (exo(?) Klenow polimerasi) completa il filo tagliato a partire dal primo nick, liberando il vecchio filone, pezzo dopo pezzo, fino a raggiungere il secondo nick. Il DNA appena completato viene ripetutamente intaccato e completato dal complesso della nickasi. I brevi filamenti singoli rilasciati da questo processo diventano il punto di partenza per un'ulteriore amplificazione in un secondo ciclo. Oltre al complesso della nickasi e alla polimerasi, le uniche cose richieste sono due primer adatti come punti di partenza per le copie.
I test con un frammento di DNA genomico batterico hanno dimostrato che la sequenza target è stata riconosciuta e amplificata con precisione. In un volume di 20 μl, è stato possibile rilevare una singola molecola. Le differenze di un singolo nucleotide all'interno di un gene potrebbero essere rilevate con elevata specificità.