Credito:Università di Leiden
Le catene proteiche tipicamente si piegano per funzionare. Il ripiegamento è un processo complesso e, se eseguito correttamente, porta a un'unica topologia di ripiegamento funzionale per una determinata catena proteica. Sono possibili anche altre topologie, ma spesso non funzionali o tossiche. Queste proteine mal ripiegate vengono quindi dispiegate e successivamente ripiegate nella topologia di piega corretta; altrimenti, vanno incontro a degrado.
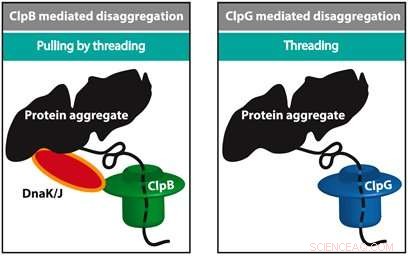
Diverse macchine tra cui ClpB e ClpG sono responsabili dello sviluppo di una proteina ripiegata. ClpB lavora a stretto contatto con HSP70 (DnaK) e HSP40 (DnaJ) e utilizza l'energia per dispiegare una catena mentre ClpG non dipende da HSP70. Una domanda importante è perché le celle sono dotate di diversi tipi di macchine e cosa determina l'efficienza dello spiegamento. Alireza Mashaghi e il suo team della LACDR/Università di Leiden hanno risolto questo enigma monitorando lo sviluppo di modelli di catene mal ripiegate a livello di singola molecola. Sono stati confrontati tre approcci in fase di sviluppo, vale a dire, infilarsi in un poro, tirando dalle estremità, e tirando per filettatura.
I risultati di questa analisi, che sono pubblicati il 25 ottobre nel Journal of Physical Chemistry B , rivelano che la topologia del circuito della catena piegata determina in modo critico il numero di percorsi e l'efficienza di svolgimento in un modo che dipende dall'approccio meccanico impiegato. Lo studio fornisce approfondimenti sui meccanismi di dispiegamento delle proteine cellulari. Questi risultati possono aiutare nella selezione di bersagli di chaperon ottimali per la farmacoterapia delle malattie da misfolding.