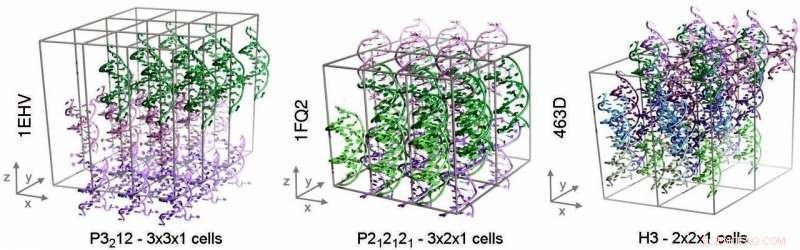
I tre sistemi cristallini esaminati in questo studio. Questi sistemi contengono (da sinistra a destra) 27, 24 e 36 DNA a doppio filamento rispettivamente. Credito:Pablo Dans Puiggròs, IRB Barcellona
Dalla nascita della biologia strutturale, La cristallografia a raggi X è stata la tecnica più utilizzata per determinare la struttura tridimensionale delle biomolecole, i composti chimici presenti negli organismi viventi. A questo proposito, la conoscenza delle interazioni tra le biomolecole con il loro ambiente cristallino e le forze molecolari che stabilizzano i cristalli servirebbe ad ottimizzare questa tecnica.
Uno studio pubblicato sulla rivista chimica e intrapreso dai ricercatori dell'Istituto per la ricerca in biomedicina (IRB Barcelona) è il primo a realizzare simulazioni stabili di cristalli di DNA. Questo risultato ha permesso agli scienziati di spiegare l'importanza degli additivi chimici usati sperimentalmente per ottenere condizioni di cristallizzazione adeguate e cristalli stabili in laboratorio.
"La prima a beneficiare di questo studio è la comunità di biofisici e fisici/chimici computazionali, che ora hanno un riferimento e protocolli chiari attraverso i quali realizzare simulazioni stabili di cristalli di DNA, " dice Pablo D. Dans, ricercatore post-dottorato presso IRB Barcelona.
Guidato da Modesto Orozco, responsabile del laboratorio di Modellazione Molecolare e Bioinformatica, lo studio presenta la descrizione atomica più dettagliata delle proprietà dei cristalli di DNA fino ad oggi.
"A lungo termine, la simulazione di diversi cristalli ottenuti in una serie di condizioni sperimentali dovrebbe consentirci di anticipare e prevedere l'effetto di un dato additivo chimico, servendo così a guidare i cristallografi nei loro esperimenti e riducendo notevolmente i costi e i tempi necessari per ottenere i cristalli, "dice Modesto Orozco, il cui laboratorio è un riferimento internazionale nel calcolo e nella simulazione bimolecolare.