Credito:Wiley
L'aggiunta e la rimozione di gruppi metilici sul DNA svolge un ruolo importante nella regolazione genica. Per studiare più precisamente questi meccanismi, un team tedesco ha sviluppato un nuovo metodo mediante il quale specifici siti di metilazione possono essere bloccati e poi sbloccati in un preciso momento mediante irradiazione con luce (photocaging). Come riportato sulla rivista Angewandte Chemie , il reggente richiesto è prodotto enzimaticamente, sul posto.
Sebbene abbiano un aspetto molto diverso e svolgano funzioni completamente diverse, tutte le cellule del nostro corpo hanno DNA identico. Però, non usano gli stessi geni. Alcuni geni sono accesi e altri spenti, a seconda del tipo di cellula e del momento. Gli "interruttori" sono cambiamenti chimici negli elementi costitutivi del DNA. Questi cambiamenti sono chiamati modificazioni epigenetiche. Un meccanismo di regolazione significativo è la metilazione e la demetilazione, che significa l'attaccamento e la rimozione di un gruppo metilico (-CH 3 ). I modelli di metilazione delle cellule tumorali, Per esempio, differiscono dalle cellule sane. Durante una metilazione, enzimi noti come metil transferasi (MTasi) trasferiscono un gruppo metilico da S-adenosil- l -metionina (AdoMet) alla molecola bersaglio.
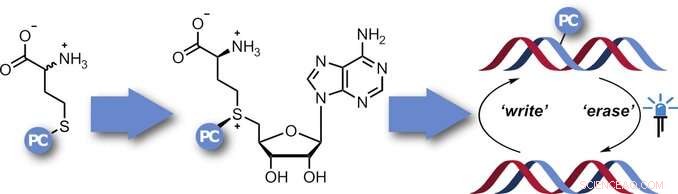
Per studiare più da vicino lo scopo e la funzione di questo regolamento e determinare i modelli di metilazione, sarebbe utile disporre di "strumenti" per inibire specificamente la metilazione in posizioni mirate e quindi rimuovere l'inibizione in un momento definito. A tal fine, un team guidato da Andrea Rentmeister ha scelto di utilizzare un metodo noto come photocaging. In questo metodo, una "fotocellula" è una molecola che si sfalda all'irradiazione, come un gruppo 2-nitrobenzile. La gabbia prima blocca la posizione di destinazione, quindi l'irradiazione mirata con la luce funge da 'interruttore' per rimuovere il blocco.
L'idea era di dotare gli analoghi di AdoMet di una fotocellula che viene poi trasferita ai siti di metilazione. Però, Gli analoghi di AdoMet si decompongono in soluzioni acquose e non possono entrare nelle cellule. Perciò, il team dell'Università di Münster voleva produrli in loco. Nel corpo, AdoMet è prodotto dall'aminoacido metionina attraverso l'azione dell'enzima, metionina adenosil transferasi (MAT). La sintesi degli analoghi di AdoMet richiede metionina con una fotocellula nitrobenzilica attaccata e un MAT in grado di utilizzare un substrato così alterato. A partire da un enzima MAT di un organismo unicellulare (Cryptosporidium hominis), i ricercatori sono stati in grado di modificare con attenzione gli amminoacidi specifici nell'enzima per aumentare le dimensioni della sua cavità di legame idrofobico in modo che potesse contenere il gruppo nitrobenzilico. Un'analisi della struttura cristallina ha mostrato che l'analogo di ADoMet è legato nella cavità di questo MAT di fotocellula (PC-MAT). Sulla base di queste informazioni, il team ha anche prodotto un secondo PC-MAT basato su un enzima termostabile MAT dell'archeone Methanocaldococcus jannaschii.
Entrambi questi PC-MAT sono compatibili con DNA e RNA MTasi e hanno permesso di attaccare fotogabbie a tutti i siti di metilazione naturale di un DNA plasmidico. L'irradiazione con la luce ha rimosso il blocco.