Sebbene l'E. coli sia uno degli organismi più studiati, la funzione del 30% delle proteine che compongono l'E. coli non è stata ancora chiaramente rivelata. Per questo, è stata utilizzata un'intelligenza artificiale per scoprire 464 tipi di enzimi dalle proteine sconosciute, e i ricercatori hanno poi verificato le previsioni di tre tipi di proteine che sono state identificate con successo attraverso un test enzimatico in vitro.
Un gruppo di ricerca congiunto, comprendente Gi Bae Kim, Ji Yeon Kim, il dottor Jong An Lee e l'illustre professore Sang Yup Lee del Dipartimento di ingegneria chimica e biomolecolare del KAIST, e il dottor Charles J. Norsigian e il professor Bernhard O. Palsson del il Dipartimento di Bioingegneria dell'UCSD, ha sviluppato DeepECtransformer, un'intelligenza artificiale in grado di prevedere le funzioni degli enzimi dalla sequenza proteica. Inoltre, il team ha creato un sistema di previsione utilizzando l'intelligenza artificiale per identificare in modo rapido e accurato la funzione dell'enzima.
Il lavoro del team è descritto nell’articolo intitolato “Annotazione funzionale di geni codificanti enzimi utilizzando l’apprendimento profondo con strati trasformatori”. L'articolo è stato pubblicato il 14 novembre su Nature Communications .
Gli enzimi sono proteine che catalizzano le reazioni biologiche e identificare la funzione di ciascun enzima è essenziale per comprendere le varie reazioni chimiche che esistono negli organismi viventi e le caratteristiche metaboliche di tali organismi.
Il numero EC (Enzyme Commission) è un sistema di classificazione della funzione enzimatica progettato dall'Unione internazionale di biochimica e biologia molecolare e, per comprendere le caratteristiche metaboliche di vari organismi, è necessario sviluppare una tecnologia in grado di analizzare rapidamente enzimi e numeri EC degli enzimi presenti nel genoma.
Sono state sviluppate varie metodologie basate sull'apprendimento profondo per analizzare le caratteristiche delle sequenze biologiche, inclusa la previsione della funzione proteica, ma la maggior parte di esse presenta il problema di una scatola nera, in cui il processo di inferenza dell'IA non può essere interpretato.
Sono stati segnalati anche vari sistemi di previsione che utilizzano l'intelligenza artificiale per la previsione della funzione enzimatica, ma non risolvono questo problema della scatola nera o non possono interpretare il processo di ragionamento a un livello a grana fine (ad esempio, il livello dei residui di amminoacidi nella sequenza dell'enzima ).
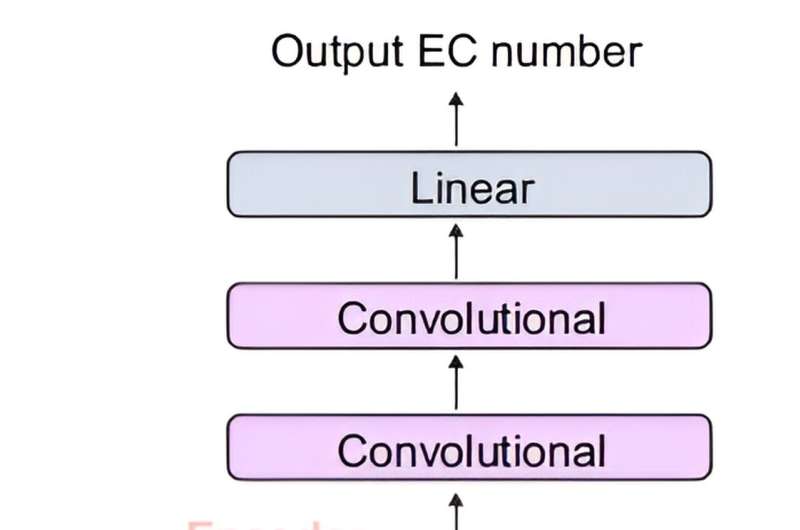
Il team congiunto ha sviluppato DeepECtransformer, un'intelligenza artificiale che utilizza l'apprendimento profondo e un modulo di analisi dell'omologia proteica per prevedere la funzione enzimatica di una determinata sequenza proteica.
Per comprendere meglio le caratteristiche delle sequenze proteiche, l'architettura del trasformatore, comunemente utilizzata nell'elaborazione del linguaggio naturale, è stata inoltre utilizzata per estrarre importanti caratteristiche sulle funzioni enzimatiche nel contesto dell'intera sequenza proteica, il che ha consentito al team di prevedere con precisione l'EC numero dell'enzima Il DeepECtransformer sviluppato può prevedere un totale di 5360 numeri EC.
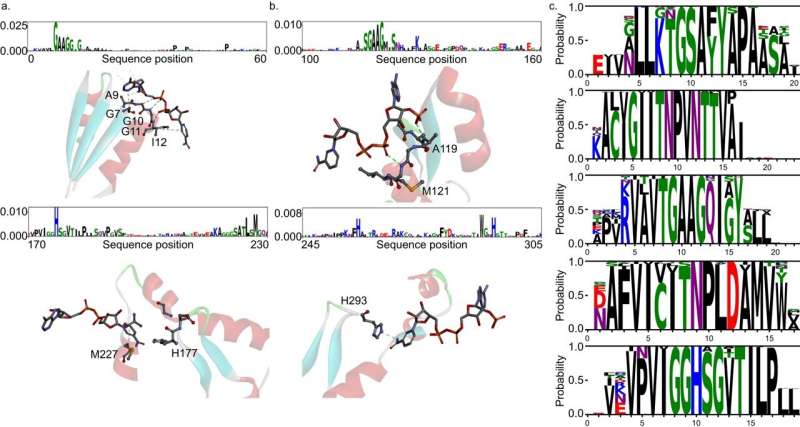
Il team congiunto ha analizzato ulteriormente l’architettura del trasformatore per comprendere il processo di inferenza di DeepECtransformer e ha scoperto che nel processo di inferenza, l’intelligenza artificiale utilizza informazioni sui siti attivi catalitici e/o sui siti di legame dei cofattori che sono importanti per la funzione enzimatica. Analizzando la scatola nera di DeepECtransformer, è stato confermato che l'intelligenza artificiale è stata in grado di identificare da sola le caratteristiche importanti per la funzione enzimatica durante il processo di apprendimento.
"Utilizzando il sistema di previsione che abbiamo sviluppato, siamo stati in grado di prevedere le funzioni degli enzimi che non erano ancora stati identificati e di verificarli sperimentalmente", ha affermato Gi Bae Kim, il primo autore dell'articolo.
"Utilizzando DeepECtransformer per identificare enzimi precedentemente sconosciuti negli organismi viventi, saremo in grado di analizzare in modo più accurato vari aspetti coinvolti nei processi metabolici degli organismi, come gli enzimi necessari per biosintetizzare vari composti utili o gli enzimi necessari per biodegradare la plastica", ha aggiunto.
"DeepECtransformer, che prevede in modo rapido e accurato le funzioni degli enzimi, è una tecnologia chiave nella genomica funzionale, poiché ci consente di analizzare la funzione di interi enzimi a livello di sistema", ha affermato il professor Sang Yup Lee.
Ha aggiunto:"Saremo in grado di usarlo per sviluppare fabbriche microbiche ecologiche basate su modelli metabolici completi su scala genomica, riducendo potenzialmente al minimo le informazioni mancanti sul metabolismo."
Ulteriori informazioni: Gi Bae Kim et al, Annotazione funzionale di geni codificanti enzimi utilizzando l'apprendimento profondo con strati trasformatori, Nature Communications (2023). DOI:10.1038/s41467-023-43216-z
Informazioni sul giornale: Comunicazioni sulla natura
Fornito dal Korea Advanced Institute of Science and Technology (KAIST)