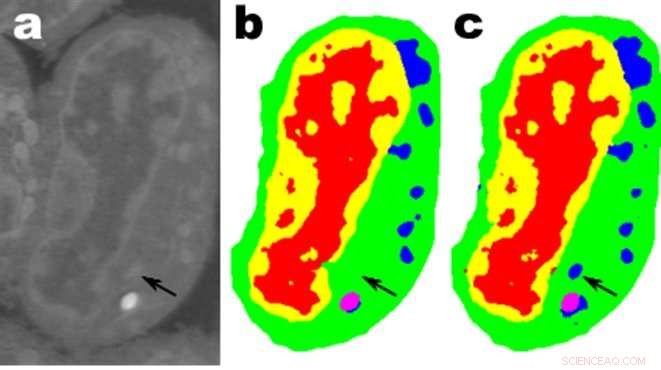
Immagini di una fetta di cellule linfoblastoidi di topo; un. sono i dati grezzi, b è la segmentazione manuale corrispondente e c è l'output di una rete MS-D con 100 livelli. Credito:dati di A. Ekman e C. Larabell, Centro nazionale per la tomografia a raggi X.
I matematici del Lawrence Berkeley National Laboratory (Berkeley Lab) del Dipartimento dell'energia hanno sviluppato un nuovo approccio all'apprendimento automatico finalizzato ai dati di imaging sperimentali. Piuttosto che fare affidamento sulle decine o centinaia di migliaia di immagini utilizzate dai tipici metodi di apprendimento automatico, questo nuovo approccio "impara" molto più rapidamente e richiede molte meno immagini.
Daniël Pelt e James Sethian del Center for Advanced Mathematics for Energy Research Applications (CAMERA) del Berkeley Lab hanno ribaltato la consueta prospettiva di apprendimento automatico sviluppando quella che chiamano una "rete neurale a convoluzione densa a scala mista (MS-D)" che richiede molti meno parametri rispetto ai metodi tradizionali, converge rapidamente, e ha la capacità di "imparare" da un set di allenamento notevolmente ridotto. Il loro approccio è già utilizzato per estrarre la struttura biologica dalle immagini cellulari, ed è pronta a fornire un nuovo importante strumento di calcolo per analizzare i dati in un'ampia gamma di aree di ricerca.
Poiché le strutture sperimentali generano immagini a risoluzione più elevata a velocità più elevate, gli scienziati possono avere difficoltà a gestire e analizzare i dati risultanti, cosa che spesso viene eseguita scrupolosamente a mano. Nel 2014, Sethian ha fondato CAMERA al Berkeley Lab come sistema integrato, centro interdisciplinare per sviluppare e fornire nuova matematica fondamentale necessaria per capitalizzare le indagini sperimentali presso le strutture degli utenti dell'Office of Science del DOE. CAMERA fa parte della Divisione di Ricerca Computazionale del laboratorio.
"In molte applicazioni scientifiche, è necessario un enorme lavoro manuale per annotare e contrassegnare le immagini:possono essere necessarie settimane per produrre una manciata di immagini accuratamente delineate, "disse Sethian, che è anche professore di matematica all'Università della California, Berkeley. "Il nostro obiettivo era sviluppare una tecnica che apprendesse da un set di dati molto piccolo".
I dettagli dell'algoritmo sono stati pubblicati il 26 dicembre 2017 in un articolo nel Atti dell'Accademia Nazionale delle Scienze .
"La svolta è derivata dalla consapevolezza che il consueto downscaling e upscaling che acquisiscono caratteristiche a varie scale di immagine potrebbe essere sostituito da circonvoluzioni matematiche che gestiscono più scale all'interno di un singolo strato, " disse Pelt, che è anche membro del Computational Imaging Group presso il Centrum Wiskunde &Informatica, l'istituto nazionale di ricerca per la matematica e l'informatica nei Paesi Bassi.
Per rendere l'algoritmo accessibile a un'ampia gamma di ricercatori, un team di Berkeley guidato da Olivia Jain e Simon Mo ha costruito un portale web "Segmenting Labeled Image Data Engine (SlideCAM)" come parte della suite di strumenti CAMERA per strutture sperimentali DOE.

Immagini tomografiche di un mini-composito fibrorinforzato, ricostruito utilizzando 1024 proiezioni (a) e 120 proiezioni (b). In (c), viene mostrato l'output di una rete MS-D con l'immagine (b) come input. Una piccola regione indicata da un quadrato rosso viene mostrata ingrandita nell'angolo in basso a destra di ogni immagine. Credito:Daniël Pelt e James Sethian, Berkeley Lab
Un'applicazione promettente è la comprensione della struttura interna delle cellule biologiche e un progetto in cui il metodo MS-D di Pelt e Sethian aveva bisogno solo dei dati di sette cellule per determinare la struttura cellulare.
"Nel nostro laboratorio, stiamo lavorando per capire come la struttura e la morfologia delle cellule influenzino o controllino il comportamento delle cellule. Trascorriamo innumerevoli ore a segmentare manualmente le cellule per estrarre la struttura, e identificare, Per esempio, differenze tra cellule sane e cellule malate, " ha detto Carolyn Larabell, Direttore del National Center for X-ray Tomography e Professore presso la University of California San Francisco School of Medicine. "Questo nuovo approccio ha il potenziale per trasformare radicalmente la nostra capacità di comprendere le malattie, ed è uno strumento chiave nel nostro nuovo progetto sponsorizzato da Chan-Zuckerberg per stabilire un atlante delle cellule umane, una collaborazione globale per mappare e caratterizzare tutte le cellule in un corpo umano sano".
Ottenere più scienza da meno dati
Le immagini sono ovunque. Smartphone e sensori hanno prodotto un tesoro di immagini, molti etichettati con informazioni pertinenti che identificano il contenuto. Utilizzando questo vasto database di immagini con riferimenti incrociati, le reti neurali convoluzionali e altri metodi di apprendimento automatico hanno rivoluzionato la nostra capacità di identificare rapidamente immagini naturali che assomigliano a quelle viste e catalogate in precedenza.
Questi metodi "imparano" mettendo a punto un insieme incredibilmente ampio di parametri interni nascosti, guidato da milioni di immagini taggate, e che richiedono grandi quantità di tempo del supercomputer. Ma cosa succede se non hai così tante immagini taggate? In molti campi, un tale database è un lusso irraggiungibile. I biologi registrano le immagini delle cellule e ne delineano accuratamente i bordi e la struttura a mano:non è insolito che una persona passi settimane a creare un'unica immagine completamente tridimensionale. Gli scienziati dei materiali usano la ricostruzione tomografica per scrutare all'interno di rocce e materiali, e poi rimboccarsi le maniche per etichettare regioni diverse, identificazione di crepe, fratture, e vuoti a mano. I contrasti tra strutture diverse ma importanti sono spesso molto piccoli e il "rumore" nei dati può mascherare le caratteristiche e confondere i migliori algoritmi (e umani).
Queste preziose immagini curate a mano non sono affatto sufficienti per i tradizionali metodi di apprendimento automatico. Per vincere questa sfida, i matematici di CAMERA hanno affrontato il problema dell'apprendimento automatico da quantità di dati molto limitate. Cercando di fare "di più con meno, " il loro obiettivo era capire come costruire un insieme efficiente di "operatori" matematici in grado di ridurre notevolmente il numero di parametri. Questi operatori matematici potrebbero naturalmente incorporare vincoli chiave per aiutare nell'identificazione, ad esempio includendo requisiti su forme e modelli scientificamente plausibili.
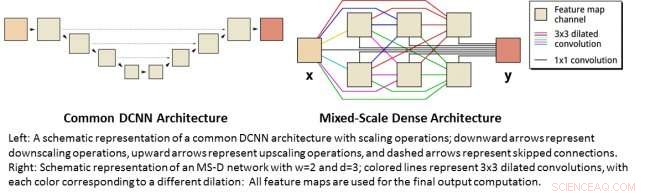
A sinistra:una rappresentazione schematica di un'architettura DCNN comune con operazioni di ridimensionamento; le frecce verso il basso rappresentano le operazioni di downscaling, le frecce verso l'alto rappresentano le operazioni di upscaling e le frecce tratteggiate rappresentano le connessioni saltate. A destra:rappresentazione schematica di una rete MS-D con w=2 e d=3; le linee colorate rappresentano circonvoluzioni dilatate 3x3, con ogni colore corrispondente a una diversa dilatazione:tutte le mappe delle caratteristiche vengono utilizzate per il calcolo dell'output finale. Credito:Daniël Pelt e James Sethian, Berkeley Lab
Reti neurali a convoluzione densa su scala mista
Molte applicazioni dell'apprendimento automatico ai problemi di imaging utilizzano reti neurali convoluzionali profonde (DCNN), in cui l'immagine in ingresso e le immagini intermedie sono convolute in un gran numero di strati successivi, consentendo alla rete di apprendere caratteristiche altamente non lineari. Per ottenere risultati accurati per problemi di elaborazione delle immagini difficili, I DCNN in genere si basano su combinazioni di operazioni e connessioni aggiuntive tra cui, Per esempio, operazioni di downscaling e upscaling per acquisire caratteristiche a varie scale di immagine. Per addestrare reti più profonde e potenti, sono spesso necessari ulteriori tipi di layer e connessioni. Finalmente, I DCNN in genere utilizzano un gran numero di immagini intermedie e parametri addestrabili, spesso più di 100 milioni, per ottenere risultati per problemi difficili.
Anziché, la nuova architettura di rete "Mixed-Scale Dense" evita molte di queste complicazioni e calcola le circonvoluzioni dilatate in sostituzione delle operazioni di ridimensionamento per catturare le caratteristiche a vari intervalli spaziali, impiegando più scale all'interno di un singolo strato, e collegando densamente tutte le immagini intermedie. Il nuovo algoritmo raggiunge risultati accurati con poche immagini e parametri intermedi, eliminando sia la necessità di ottimizzare gli iperparametri che livelli o connessioni aggiuntivi per consentire l'addestramento.
Ottenere scienza ad alta risoluzione da dati a bassa risoluzione
Una sfida diversa consiste nel produrre immagini ad alta risoluzione da input a bassa risoluzione. Come chiunque abbia provato a ingrandire una piccola foto e ha scoperto che peggiora solo quando diventa più grande, questo sembra quasi impossibile. Ma un piccolo set di immagini di addestramento elaborate con una rete densa a scala mista può fornire progressi reali. Come esempio, immagina di provare a denoising ricostruzioni tomografiche di un materiale mini-composito fibrorinforzato. In un esperimento descritto nel documento, le immagini sono state ricostruite utilizzando 1, 024 ha acquisito proiezioni di raggi X per ottenere immagini con quantità di rumore relativamente basse. Le immagini rumorose dello stesso oggetto sono state poi ottenute ricostruendo con 128 proiezioni. Gli input di allenamento erano immagini rumorose, con corrispondenti immagini silenziose utilizzate come output target durante l'allenamento. La rete addestrata è stata quindi in grado di acquisire in modo efficace dati di input rumorosi e ricostruire immagini a risoluzione più elevata.
Nuove applicazioni
Pelt e Sethian si stanno avvicinando a una serie di nuove aree, come l'analisi veloce in tempo reale delle immagini provenienti da sorgenti di luce di sincrotrone e problemi di ricostruzione nella ricostruzione biologica come per le cellule e la mappatura del cervello.
"Questi nuovi approcci sono davvero entusiasmanti, poiché consentiranno l'applicazione dell'apprendimento automatico a una varietà di problemi di imaging molto più ampia di quella attualmente possibile, " ha detto Pelt. "Riducendo la quantità di immagini di addestramento richieste e aumentando la dimensione delle immagini che possono essere elaborate, la nuova architettura può essere utilizzata per rispondere a domande importanti in molti campi di ricerca."