
Architettura del telescopio:il gestore del lavoro raccoglie le informazioni sul lavoro connettendosi al cluster di destinazione tramite il suo gestore di connessione. Il database locale del telescopio conserva le registrazioni di queste informazioni, che è reso dall'interfaccia utente in una pagina web (mobile friendly). Credito:Brito et al.
Un team di ricercatori dell'UCLA, l'Università di San Paolo, la Federal University of São Carlos e la University of Southern California hanno recentemente sviluppato uno strumento interattivo per la gestione di analisi bioinformatiche su larga scala in tempo reale e da dispositivi portatili. Questo nuovo strumento, chiamato telescopio, è stato presentato per la prima volta in un documento pre-pubblicato su arXiv .
Il team che ha sviluppato Telescope include borsisti post-dottorato della UCLA Collaboratory, un laboratorio di ricerca presso l'Istituto universitario per le bioscienze quantitative e computazionali. Lavorare presso l'UCLA Collaboratory consente a questi ricercatori di interagire con altri studenti e docenti dell'università che hanno diversi livelli di competenza in biologia computazionale. Queste interazioni sono ciò che alla fine ha ispirato il team a sviluppare uno strumento di facile utilizzo per la gestione di analisi bioinformatiche su larga scala.
"La maggior parte delle scienze della vita e degli studi biomedici oggi mirano a effetti così sottili e difficili da rilevare che grandi quantità di dati e potenza di calcolo sono diventati il nuovo status quo, " hanno detto i ricercatori a TechXplore via e-mail. "Spesso, però, i ricercatori descrivono l'eccessiva dipendenza dagli strumenti della riga di comando come ingombrante e altamente non intuitiva per coloro che non hanno una formazione computazionale formale".
Iniziative di formazione specializzata e workshop si sono rivelati particolarmente efficaci nell'incoraggiare i ricercatori a iniziare a utilizzare metodi potenti per analisi bioinformatiche su larga scala. Però, la maggior parte delle tecniche per accedere a questi metodi su strutture computazionali non sono molto ben integrate con ambienti di laboratorio umido, dove si svolgono la maggior parte degli esperimenti di biologia. Con questo in testa, i ricercatori hanno deciso di sviluppare uno strumento che consentirebbe una migliore integrazione tra gli strumenti di bioinformatica e la ricerca in laboratorio umido.
"Il telescopio è stato proposto per favorire l'integrazione e una più stretta collaborazione tra la ricerca in laboratorio umido e la bioinformatica, ridurre la curva di apprendimento tradizionalmente richiesta per eseguire analisi bioinformatiche complesse in strutture computazionali, " ha detto il team a TechXplore. "La sfida, però, garantiva che la soluzione finale avrebbe effettivamente colmato le lacune e sarebbe stata intuitiva per la maggior parte dei ricercatori".
Per presentare la loro soluzione sia ai ricercatori esperti di tecnologia che a quelli con capacità computazionali limitate, i ricercatori hanno deciso di introdurre Telescope durante l'evento II Hackathon all'inizio del 2018. Hackathon riunisce ricercatori con competenze informatiche e di programmazione, offrendo loro l'opportunità di acquisire competenze informatiche in gruppo, ad esempio, esplorando come costruire script e applicare strumenti bioinformatici ai set di dati.
"La nostra idea ha attratto sia i neofiti che volevano sporcarsi le mani lavorando direttamente con i dati il più rapidamente possibile, sia gli esperti di tecnologia che volevano solo un modo più semplice per controllare il proprio lavoro dai loro telefoni, " hanno spiegato i ricercatori. "Il telescopio è stato quindi progettato da e per la comunità".
La maggior parte dei ricercatori che effettuano esperimenti in ambienti di laboratorio umidi deve soddisfare notevoli esigenze di tempo. Il telescopio è progettato per aiutarli a gestire meglio il loro carico di lavoro, supervisione e regolazione delle analisi bioinformatiche in tempo reale.
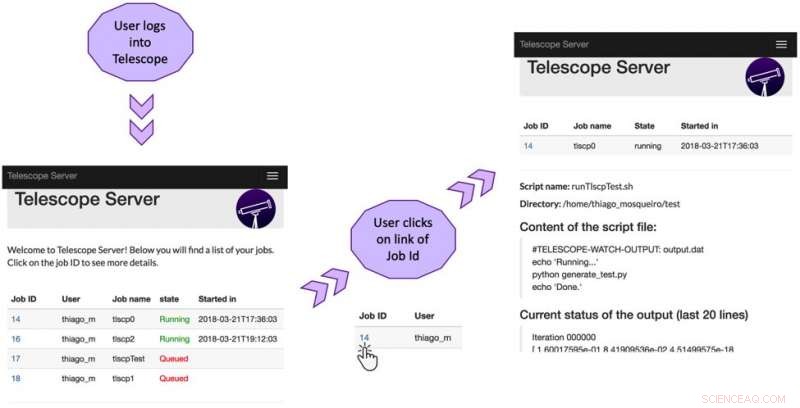
Interfaccia utente del telescopio:la prima schermata visualizza lo stato dei lavori sul cluster. La schermata successiva mostra informazioni dettagliate sul primo lavoro elencato:directory di origine, nome e contenuto del file di script, e le ultime righe dell'output dell'attività corrente. Credito:Brito et al.
Essenzialmente, Il telescopio rafforza la relazione tra gli esperimenti di laboratorio umido e gli strumenti per l'analisi di grandi quantità di dati biologici. Lo fa fornendo una piattaforma sicura e di facile utilizzo su cui gli utenti possono avviare la loro ricerca e gestire attività rilevanti per i lavori di bioinformatica in esecuzione in cluster ad alte prestazioni.
"Il telescopio sfrutta framework web comuni come Bootstrap di Twitter per fornire un'esperienza utente accattivante e supportare un'interfaccia estensibile, " hanno detto i ricercatori. "Intendendo supportare sia gli utenti nuovi che quelli esperti, Il core di Telescope gestisce e archivia le chiavi di accesso degli utenti utilizzando gli standard del settore."
Le chiavi di accesso memorizzate da Telescope vengono utilizzate per connettere cluster computazionali ad alte prestazioni tramite una shell sicura al fine di ricevere gli ultimi aggiornamenti sullo stato dei lavori ed emettere nuovi comandi ogni volta che è necessario (ad es. cancellazione di vecchi compiti, ecc.). Il telescopio consente agli scienziati del laboratorio umido di monitorare lo stato del loro lavoro e di accedere facilmente ai risultati preliminari delle loro analisi bioinformatiche direttamente sui loro telefoni cellulari. Ciò significa che possono anche identificare in anticipo potenziali problemi con le loro analisi e annullarli senza dover utilizzare un computer.
"Rispetto al solito approccio all'utilizzo delle righe di comando sui terminali Linux, Il telescopio è uno strumento più intuitivo e interattivo, " hanno spiegato i ricercatori. "Per esempio, condividere i risultati dell'analisi è facile come condividere un link, come faresti su Twitter o Facebook. Ciò nonostante, riteniamo che il nostro risultato più significativo sia stato l'impegno con la comunità delle scienze della vita tradizionalmente non computazionale all'inizio dello sviluppo".
Questo nuovo strumento per la gestione delle analisi bioinformatiche ha tenuto in stretta considerazione le esigenze dei ricercatori in biologia. Infatti, il team ha approfittato dell'evento Hackathon per raccogliere input e feedback da esperti di biologia con vari gradi di competenza ed esperienza IT utilizzando strumenti di analisi computazionale.
"Abbiamo raccolto input da utenti computazionali alle prime armi, poiché volevamo creare uno strumento che affrontasse i punti più impegnativi dell'utilizzo di righe di comando non intuitive, " hanno detto i ricercatori. "Abbiamo quindi sviluppato un'interfaccia web semplice e minimale che può essere eseguita su vari dispositivi, compresi i telefoni cellulari, con impostazione zero. La sfida era quella di scambiare semplicità con sicurezza, e abbiamo raggiunto un buon equilibrio utilizzando protocolli standard del settore, come memorizzare le chiavi usando PBDKS."
Lo strumento sviluppato da questo gruppo di ricercatori, che è ora disponibile su GitHub, possono assistere gli esperti di biologia nel loro lavoro, aiutandoli a gestire le proprie analisi bioinformatiche in modo più semplice e intuitivo. Oltre a consentire ai ricercatori biomedici di accedere alla potenza di grandi strutture computazionali dai loro dispositivi portatili, Telescope è un software personalizzabile ed estensibile. Ciò significa che gli utenti possono aggiungere nuove funzionalità o contribuire a svilupparlo ulteriormente.
"Il nostro progetto sarà ora mantenuto dal Mangul Lab presso la USC come software open-source, " hanno detto i ricercatori. "Stiamo anche progettando di integrare Telescope nella maggior parte delle pipeline di analisi del nostro laboratorio al fine di valutare ulteriormente l'esperienza dell'utente. Come passo successivo, questi test saranno estesi a un sondaggio di beta tester di una comunità più ampia. Il nostro obiettivo principale è quello di implementare Telescope per gli utenti del cluster ad alte prestazioni di USC in futuro. Durante questo processo, il laboratorio Mangul prospererà per impegnarsi con la comunità per guidare l'adozione e garantire che Telescope rimanga rilevante".
© 2019 Scienza X Rete