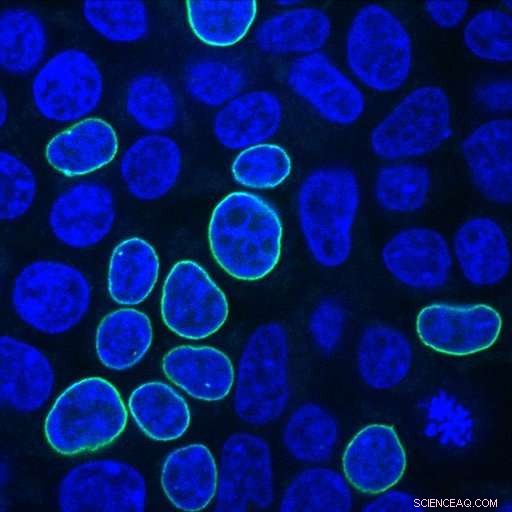
Le cellule renali embrionali umane diventano verdi dopo la riparazione di una rottura del DNA indotta da CRISPR con un frammento di PCR che codifica una proteina fluorescente e bracci di omologia con 33 nucleotidi. Credito:Alexandre Paix
Gli scienziati della Johns Hopkins hanno sviluppato un metodo semplificato e relative "regole" di efficienza per introdurre nuove sequenze di DNA nelle cellule dopo aver utilizzato lo strumento di taglio genetico noto come CRISPR. Gli scienziati dicono che il metodo, che hanno basato su test con embrioni di topo e migliaia di cellule umane, potrebbe migliorare la coerenza e l'efficienza dell'editing del genoma.
Il nuovo metodo e il suo sviluppo sono descritti online nel 28 novembre nel Atti dell'Accademia Nazionale delle Scienze .
"CRISPR è uno strumento per aiutare gli scienziati a modificare il genoma, prevedere l'esito di determinati tratti e studiarli, ma lo strumento stesso crea solo rotture nel genoma. Non controlla come una nuova sequenza di DNA viene inserita nel genoma, "dice Geraldine Seydoux, dottorato di ricerca, l'Huntington Sheldon Professor in Medical Discovery nel Dipartimento di Biologia Molecolare e Genetica e vice preside per la ricerca di base presso la Johns Hopkins University School of Medicine, e un investigatore con l'Howard Hughes Medical Institute.
"Abbiamo deciso di studiare come le cellule riparano le rotture indotte da CRISPR con l'obiettivo di utilizzare il processo di riparazione naturale del DNA della cellula per introdurre nuove sequenze nel genoma. Siamo rimasti sorpresi nello scoprire che le cellule copieranno prontamente sequenze da DNA estraneo per riparare le rotture del DNA. , fintanto che i DNA estranei sono lineari, " Seydoux aggiunge. "Studiando come i frammenti di DNA estraneo vengono copiati durante il processo di riparazione, abbiamo escogitato alcune semplici regole per rendere l'editing del genoma il più efficiente possibile, ottimizzare lo strumento, e fallo con fiducia».
CRISPR, che sta per brevi ripetizioni palindromiche a cluster regolarmente interspaziate, ha guadagnato popolarità tra gli scienziati negli ultimi cinque anni come strumento per tagliare in modo efficiente il DNA. È stato adattato per l'uso nelle cellule di mammifero da un processo di difesa virale naturale nelle cellule batteriche che comporta la creazione di tagli letali nel DNA virale. Essenzialmente, lo strumento è un insieme semplificato di "forbici" molecolari.
La convinzione prevalente, tra gli scienziati, è che le cellule riparano le rotture del DNA inserendo un insieme casuale di nucleotidi, gli elementi costitutivi chimici del DNA. Questo di solito distrugge qualsiasi gene che si trova nel punto in cui il DNA è rotto.
È anche ben noto agli scienziati che, occasionalmente, le cellule utilizzano una fonte diversa:una sequenza di un altro pezzo di DNA, o DNA "donatore" per sigillare la rottura nel DNA. Però, la nuova sequenza "donatore" non può essere inserita da sola in uno spazio vuoto nel genoma.
Anziché, il nuovo DNA del donatore ha bisogno di una specie di nastro a ciascuna estremità per aiutarlo a rimanere all'interno dello spazio creato dal taglio. Gli scienziati si riferiscono a questo nastro come alle braccia di "omologia" del DNA del donatore.
I bracci di omologia sono costituiti da nucleotidi che si sovrappongono alle porzioni intatte del DNA con il codice genetico corrispondente. Questo aiuta il DNA del donatore ad "attaccarsi" al DNA intatto.
Ancora, gli scienziati consideravano l'uso del DNA del donatore un modo inefficiente per riparare il genoma, supponendo che richiedesse lunghe braccia di omologia, soprattutto quando si inserisce una lunga sequenza di DNA, e DNA a filamento singolo o circolare, difficili da preparare in pezzature lunghe.
Man mano che gli scienziati acquisivano maggiore esperienza con CRISPR, Seydoux dice, "Sono sorte domande sulle regole di progettazione ottimale per il DNA del donatore e sulla lunghezza dei bracci di omologia".
Cercando risposte a queste domande, gli scienziati della Johns Hopkins hanno inserito varie combinazioni di DNA donatore in cellule renali embrionali umane, noti per la loro capacità di crescere bene e per il loro uso frequente nella ricerca sul cancro. Gli scienziati hanno utilizzato il DNA di un donatore con un gene che codifica per una proteina fluorescente, che si illumina di verde nella membrana nucleare della cellula quando l'inserimento del gene ha successo.
Il ricercatore associato della Johns Hopkins Alexandre Paix ha scoperto che i frammenti di DNA lineare funzionano molto bene come donatori, e sono da due a cinque volte più efficienti dei DNA circolari (noti come plasmidi) nelle cellule umane. "Il DNA lineare è molto facile da preparare in laboratorio, utilizzando la PCR, "dice Paix, riferendosi agli strumenti di reazione a catena della polimerasi, che servono per amplificare il DNA.
Paix ha anche testato varie lunghezze di bracci di omologia. Ha scoperto che il punto debole per i bracci di omologia è lungo circa 35 nucleotidi, molto più breve di quanto normalmente usano gli scienziati.
Nello specifico, si è riscontrato che i bracci di omologia di lunghezza compresa tra 33 e 38 nucleotidi avevano lo stesso successo di quelli con 518 nucleotidi, ottenendo tra il 10 e il 20 percento di modifiche riuscite in condizioni ottimali. In contrasto, quando gli scienziati hanno testato bracci di omologia di 15 e 16 nucleotidi di lunghezza, le percentuali di successo dell'inserimento si sono dimezzate. Hanno ripetuto questi risultati in tre diverse posizioni nel genoma umano.
Hanno anche scoperto che la sequenza appena inserita, senza contare le armi di omologia, può essere fino a 1, 000 nucleotidi di lunghezza.
Il team ha raggiunto tassi di successo tra il 10 e il 50 percento con inserti di lunghezza compresa tra 57 e 993 nucleotidi. Le sequenze più brevi sono state inserite con maggior successo rispetto a quelle più lunghe. Per esempio, nuove sequenze che erano 57, 714 e 993 nucleotidi lunghi sono stati inseriti con successo 45,4, 23,5 e 17,9 per cento del tempo, rispettivamente. Oltre 1, 000 nucleotidi, nuovi inserti con 1, 122 e 2, 229 nucleotidi hanno avuto scarso successo, circa lo 0,5 percento delle volte. "A quelle dimensioni, diventa molto difficile introdurre la quantità di DNA del donatore necessaria per l'editing. Le cellule tendono a "soffocare" su così tanto DNA, "dice Seydoux.
Finalmente, il team ha anche scoperto che il tasso di successo dell'editing raggiunge un picco quando la nuova sequenza viene posizionata entro 30 nucleotidi dal sito di taglio CRISPR. "Oltre i 30 nucleotidi, l'inserimento non è praticabile, "dice Seydoux.
"Questi parametri dovrebbero adattarsi alla maggior parte dei geni che gli scienziati stanno cercando di modificare. In effetti, la maggior parte degli esperimenti comporta la modifica di solo due o tre nucleotidi vicino al sito di taglio CRISPR, " aggiunge Seydoux.
Il team di ricerca ha anche testato se lo stesso approccio potesse funzionare negli embrioni di topo. Utilizzando un frammento PCR con bracci di omologia a 36 nucleotidi, il team ha inserito con successo una sequenza lunga 739 nucleotidi che codifica per una proteina fluorescente in 27 su 87 (31 percento) embrioni di topo.
Il team di ricerca di Seydoux sta già utilizzando le regole di riparazione per studiare il DNA in Caenorhabditis elegans, una specie di verme, ei ricercatori stanno studiando se le regole di riparazione si applicano ad altri tipi di cellule umane.
Prima che le linee guida siano ampiamente adottate, Seydoux dice che dovrebbero essere testati in più tipi di cellule umane e altri organismi.