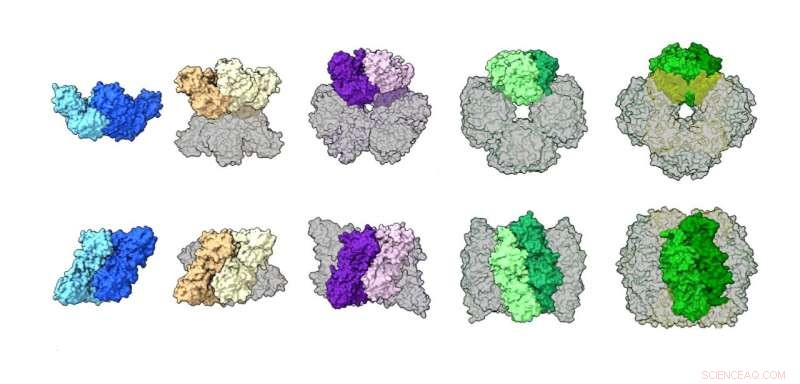
Un confronto tra assemblaggi di rubisco di specie diverse, che illustra un'ampia gamma di strutture tra cui un dimero, un tetramero, un esamero, un ottamero e un esadecamero (proteina a 16 unità). Credito:Shih Lab/Berkeley Lab
Quando pensi alle proteine, agli enzimi, alle molecole di segnalazione e ai componenti strutturali di ogni essere vivente, potresti pensare a singoli filamenti di amminoacidi, organizzati come perline su un filo. Ma quasi tutte le proteine sono costituite da più filamenti ripiegati e legati l'uno all'altro, formando complicate sovrastrutture 3D chiamate assemblaggi molecolari. Uno dei passaggi chiave per comprendere la biologia è scoprire come una proteina svolge il suo lavoro, il che richiede la conoscenza delle sue strutture fino al livello atomico.
Nel secolo scorso, gli scienziati hanno sviluppato e implementato tecnologie straordinarie come la cristallografia a raggi X e la microscopia crioelettronica per determinare la struttura delle proteine, e quindi hanno risposto a innumerevoli domande importanti. Ma un nuovo lavoro mostra che la comprensione della struttura delle proteine a volte può essere più complicata di quanto pensiamo.
Un gruppo di ricercatori del Lawrence Berkeley National Laboratory (Berkeley Lab) che studia la proteina più abbondante del mondo, un enzima coinvolto nella fotosintesi chiamato rubisco, ha mostrato come l'evoluzione può portare a una sorprendente diversità di assemblaggi molecolari che svolgono tutti lo stesso compito. I risultati, pubblicati oggi su Science Advances , rivelano la possibilità che molte delle proteine che pensavamo di conoscere esistano effettivamente in altre forme sconosciute.
Storicamente, se gli scienziati risolvessero una struttura e determinassero che una proteina è dimerica (composta da due unità), ad esempio, potrebbero presumere che proteine simili esistessero anche in forma dimerica. Ma piccole dimensioni del campione e bias di campionamento, fattori inevitabili dato che è molto difficile convertire le proteine naturalmente liquide in forme solide e cristallizzate che possono essere esaminate tramite cristallografia a raggi X – oscuravano la realtà.

Albert Lui (a sinistra) e Patrick Shih dimostrano come hanno studiato la struttura e l'evoluzione del rubisco, l'enzima che le piante usano per sfruttare la CO2 per creare zuccheri, nella Koshland Hall della UC Berkeley. Credito:Marilyn Sargent/Berkeley Lab
"È come se uscissi e vedessi qualcuno portare a spasso il cane, se non avessi mai visto un cane prima e poi vedessi un cane wiener, penseresti:'OK, ecco come sono tutti i cani.' Ma quello che devi fare è andare al parco per cani e vedere tutta la diversità dei cani che c'è lì", ha affermato l'autore principale Patrick Shih, uno scienziato della facoltà nell'area delle bioscienze e direttore della progettazione dei biosistemi vegetali presso il Joint BioEnergy Institute (JBEI). "Un takeaway da questo documento che va oltre il rubisco, a tutte le proteine, è la questione se stiamo vedendo o meno la vera gamma di strutture in natura, o se questi pregiudizi fanno sembrare che tutto assomigli a un cane wiener."
Sperando di esplorare tutti i diversi arrangiamenti di rubisco nel metaforico parco dei cani e di scoprire da dove provenissero, il laboratorio di Shih ha collaborato con esperti di biologia strutturale dell'area della bioscienza utilizzando la sorgente di luce avanzata di Berkeley Lab. Insieme, il team ha studiato un tipo di rubisco (forma II) che si trova nei batteri e un sottoinsieme di microbi fotosintetici utilizzando la cristallografia tradizionale, una tecnica in grado di ottenere una risoluzione a livello atomico, combinata con un'altra tecnica di risoluzione della struttura, lo scattering di raggi X a piccolo angolo (SAXS), che ha una risoluzione inferiore ma può scattare istantanee delle proteine nella loro forma nativa quando si trovano in miscele liquide. SAXS ha l'ulteriore vantaggio di una capacità ad alto rendimento, il che significa che può elaborare dozzine di singoli gruppi proteici in rapida successione.
Il lavoro precedente aveva dimostrato che il tipo più studiato di rubisco trovato nelle piante (forma I) prende sempre un "nucleo ottamerico" assemblato di otto grandi unità proteiche disposte con otto piccole unità, mentre si credeva che la forma II esistesse principalmente come dimero con un pochi rari esempi di esameri a sei unità. Dopo aver utilizzato queste tecniche complementari per esaminare campioni di rubisco da una vasta gamma di specie microbiche, gli autori hanno osservato che la maggior parte delle proteine di rubisco della forma II sono in realtà esameri, con l'occasionale dimero, e hanno scoperto un tetramerico mai visto prima (quattro unità) assemblaggio.
La combinazione di questi dati strutturali con le rispettive sequenze geniche codificanti le proteine ha consentito al team di eseguire la ricostruzione della sequenza ancestrale, un metodo di evoluzione molecolare computerizzato in grado di stimare l'aspetto delle proteine ancestrali in base alla sequenza e all'aspetto delle proteine moderne che si sono evolute da esse.
Albert Liu controlla alcune piastre di coltura di E. coli con i geni per formare la forma II rubisco. Credito:Albert Liu
La ricostruzione suggerisce che il gene per la forma II rubisco è cambiato nel corso della sua storia evolutiva per produrre proteine con una gamma di strutture che si trasformano in nuove forme o ritornano abbastanza facilmente a strutture più vecchie. Al contrario, nel corso dell'evoluzione, le pressioni selettive hanno portato a una serie di cambiamenti che hanno bloccato la forma I rubisco in posizione - un processo chiamato trinceramento strutturale - motivo per cui l'assieme ottamerico è l'unica disposizione che vediamo ora. Secondo gli autori, si presumeva che la maggior parte degli assiemi proteici fossero trincerati nel tempo da una pressione selettiva per affinare la loro funzione, come vediamo con la forma I rubisco. Ma questa ricerca suggerisce che l'evoluzione può anche favorire le proteine flessibili.
"La grande scoperta di questo documento è che c'è molta plasticità strutturale", ha detto Shih, che è anche assistente professore alla UC Berkeley. "Le proteine possono essere molto più flessibili, sul campo, di quanto credessimo."
Dopo aver completato la ricostruzione della sequenza ancestrale, il team ha condotto esperimenti mutazionali per vedere come l'alterazione dell'assemblaggio del rubisco, in questo caso la rottura di un esamero in un dimero, influisse sull'attività dell'enzima. Inaspettatamente, questa mutazione indotta ha prodotto una forma di rubisco che utilizza meglio la sua molecola bersaglio, CO2 . Tutto il rubisco presente in natura lega spesso O2 di dimensioni simili molecola accidentalmente, riducendo la produttività dell'enzima. C'è un grande interesse nel modificare geneticamente il rubisco nelle specie vegetali agricole per aumentare l'affinità della proteina per la CO2 , al fine di produrre colture più produttive ed efficienti sotto il profilo delle risorse. Tuttavia, c'è stata molta attenzione sul sito attivo della proteina, la regione della proteina in cui CO2 o O2 vincolare.
"Questa è un'intuizione interessante per noi perché suggerisce che per avere risultati più fruttuosi nell'ingegneria del rubisco, non possiamo semplicemente guardare alla risposta più semplice, la regione dell'enzima che interagisce effettivamente con la CO2 ", ha detto il primo autore Albert Liu, uno studente laureato nel laboratorio di Shih. "Forse ci sono mutazioni al di fuori di quel sito attivo che effettivamente partecipano a questa attività e possono potenzialmente modificare la funzione delle proteine nel modo che vogliamo. Quindi è qualcosa che apre davvero le porte a future vie di ricerca."
Il coautore Paul Adams, Associate Laboratory Director for Biosciences e Vice President for Technology presso JBEI ha aggiunto:"Il mix di tecniche impiegate e la natura interdisciplinare del team è stata una vera chiave del successo. Il lavoro mette in evidenza il potere di combinare dati genomici e metodi di biologia strutturale per studiare uno dei problemi più importanti della biologia e raggiungere alcune conclusioni inaspettate". + Esplora ulteriormente