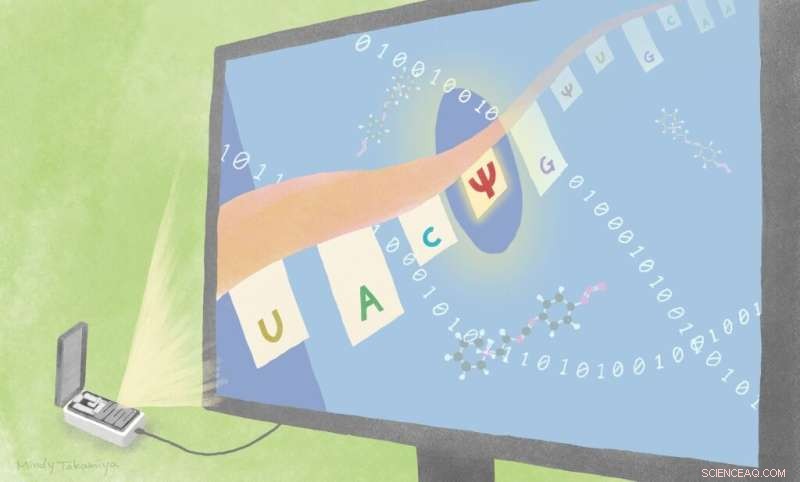
Gli scienziati hanno trovato due nuovi modi per identificare le sostituzioni dall'uracile alla pseudouridina, combinando la tecnologia di sequenziamento esistente con approcci diversi. Uno è con calcolo basato su algoritmi e uno è con una sonda chimica CMC. Credito:Mindy Takamiya/iCeMS dell'Università di Kyoto
Due nuovi approcci potrebbero aiutare gli scienziati a utilizzare la tecnologia di sequenziamento esistente per distinguere meglio i cambiamenti dell'RNA che influenzano il modo in cui viene letto il codice genetico.
Gli scienziati dell'Università di Kyoto si stanno avvicinando alla ricerca di modi per identificare i cambiamenti nelle sequenze di RNA che influiscono sulla formazione di proteine e possono causare malattie. Il loro approccio, pubblicato sulla rivista Genomics , utilizza algoritmi di probabilità insieme a un dispositivo di sequenziamento tascabile già disponibile.
"Le modifiche che si trovano in tutti i tipi di RNA biologico influenzano la regolazione genica, che alla fine decide come funzionano le diverse cellule nel nostro corpo", spiega Ganesh Pandian Namasivayam dell'Istituto per la scienza integrata dei materiali cellulari (WPI-iCeMS) dell'Università di Kyoto. "Anomalie in queste modifiche possono portare a malattie gravi, come diabete, disturbi del neurosviluppo e cancro. Sapere come e dove si trovano queste modificazioni dell'RNA è di primaria importanza da un punto di vista clinico", aggiunge Soundhar Ramaswamy, il primo autore dello studio.
Esistono già modi per identificare le modifiche dell'RNA, ma sono insufficienti. Gli approcci biofisici come la cromatografia e la spettrometria di massa possono elaborare solo piccole quantità di RNA alla volta. I metodi di sequenziamento ad alto rendimento, che possono elaborare grandi quantità di RNA, implicano una laboriosa preparazione del campione, non possono mappare più modifiche contemporaneamente e sono soggetti a errori.
Namasivayam, Hiroshi Sugiyama e colleghi dell'Università di Kyoto hanno testato e trovato due approcci che possono distinguere con successo una modificazione dell'RNA ben nota e abbondante che implica la sostituzione del nucleotide base dell'uracile con un altro chiamato pseudouridina.
Simile al DNA, l'RNA è formato da un filamento di diverse combinazioni di quattro diverse basi nucleotidiche:uracile, citosina, adenina e guanina. Il modo in cui queste basi sono disposte determina il codice che segnala quale proteina deve essere prodotta. Quando la pseudouridina sostituisce l'uracile nella spina dorsale dell'RNA, può portare a un aumento della produzione di proteine o alla modifica del codice da uno che segnala l'interruzione della traduzione delle informazioni a uno che segnala la formazione di aminoacidi.
L'approccio del team prevede l'utilizzo di una piattaforma di sequenziamento dell'RNA diretto già disponibile sviluppata da Oxford Nanopore Technologies. In questa piattaforma, i filamenti di RNA passano attraverso minuscoli pori in una membrana. Le interruzioni sono causate nella corrente che si muove attraverso la membrana a seconda dell'ordine delle diverse basi dell'RNA. Ciò consente agli scienziati di "leggere" la sequenza. Ma gli scienziati che utilizzano questo approccio spesso trovano difficile distinguere i diversi tipi di modifiche l'uno dall'altro.
Shubham Mishra, uno dei primi autori di questo studio, ha sviluppato algoritmi per identificare un'alta probabilità di esistenza di una sostituzione della pseudouridina rispetto alla possibilità che si trattasse di un diverso tipo di cambiamento di base.
Una delle loro strategie confronta brevi sequenze di RNA di cinque basi nucleotidiche in cui uracile, pseudouridina o citosina sono circondate su entrambi i lati dalle stesse basi. Le letture passano quindi attraverso algoritmi che calcolano la probabilità che la base media sia una delle tre. Hanno usato la loro strategia, chiamata Indo-Compare (Indo-C), su sequenze di RNA ingegnerizzate e poi su lievito e RNA umano e hanno scoperto che era efficace nel distinguere le sostituzioni di pseudouridina dalle altre.
Sono stati anche in grado di identificare le sostituzioni di pseudouridina mescolando una sonda chimica con campioni di RNA, che poi si attacca selettivamente ad essi. Ciò ha modificato le letture della sequenza in modo da identificare la modifica.
"Riteniamo che il nostro lavoro renderà i metodi basati sul sequenziamento dei nanopori meno laboriosi per rilevare le modifiche dell'RNA e più in grado di caratterizzare l'impatto di queste modifiche sullo sviluppo e sulla malattia", afferma Namasivayam.
Il team poi mira a ottimizzare l'uso di entrambi gli approcci insieme per identificare più accuratamente le modifiche dell'RNA e del DNA. Ciò comporterà la fabbricazione di nuove sonde chimiche che corrispondono a cambiamenti specifici. Hanno inoltre in programma di sviluppare ulteriormente algoritmi avanzati di apprendimento automatico che integrano gli approcci di sequenziamento diretto dell'RNA basati su sonde chimiche. + Esplora ulteriormente