Quando si tenta di misurare strutture molecolari con precisione nanometrica, ogni minimo rumore si manifesta nei dati:qualcuno che passa davanti al microscopio, minuscole vibrazioni nell'edificio e persino il traffico all'esterno. Una nuova tecnica di elaborazione rimuove il rumore dai dati del microscopio ottico in tempo reale, consentendo agli scienziati di tracciare le singole molecole con una precisione 10 volte maggiore rispetto a quanto fosse possibile prima.
Un team di ricercatori di bioingegneria dell’Università dell’Illinois Urbana-Champaign ha introdotto un algoritmo chiamato massimizzazione dell’intersezione adattiva, o AIM, che rimuove il rumore ad alta frequenza dai dati del microscopio ottico a super risoluzione molto più velocemente rispetto ai metodi standard e si traduce in risoluzioni di immagine molto più elevate. .
L'algoritmo consentirà agli scienziati di studiare i sistemi chimici e biologici in modo molto più semplice e preciso di quanto fosse possibile prima. Questa ricerca è stata pubblicata sulla rivista Science Advances.
"All'inizio, volevamo solo sviluppare un algoritmo veloce perché il nostro laboratorio produce troppi dati per essere gestiti dagli algoritmi tradizionali, ma abbiamo scoperto che AIM può anche raggiungere una precisione sub-nanometrica, cosa inaudita nel nostro campo", ha affermato Hongqiang Ma , professore di ricerca di bioingegneria e autore principale dello studio. "Inoltre, non richiede un'enorme potenza di calcolo come gli strumenti tradizionali. Può essere eseguito su un laptop. Vogliamo renderlo uno strumento plug-and-play per tutti gli utenti di microscopi."
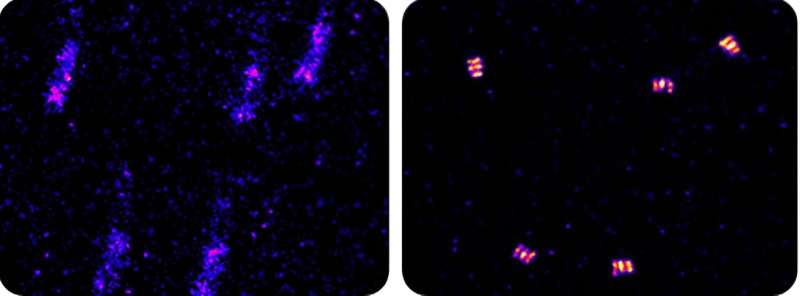
Negli ultimi decenni, la tecnica della microscopia di localizzazione di singole molecole ha consentito agli scienziati di visualizzare strutture su scala molecolare, superando quella che si pensava fosse una limitazione fondamentale dei microscopi ottici. Tuttavia, nella pratica è limitato da un rumore incontrollabile, o "deriva", che sostanzialmente offusca le immagini e impedisce alla microscopia a super risoluzione di raggiungere la risoluzione massima.
"La localizzazione di singole molecole in realtà utilizza uno strumento abbastanza semplice, ma la parte difficile che ha un vero impatto sulla risoluzione dell'immagine è la deriva", ha affermato Yang Liu, professore di bioingegneria e responsabile del progetto. "Molti ricercatori rimuovono solo la deriva a bassa frequenza. La rimozione della deriva ad alta frequenza, ovvero le minuscole vibrazioni causate dal rumore ambientale, è un lavoro intensivo dal punto di vista computazionale e richiede grandi quantità di tempo e risorse."
I metodi standard per rimuovere la deriva si basano sulle correlazioni matematiche tra i fotogrammi dell'immagine. Secondo Liu, i microscopi nel suo laboratorio generano un volume di dati di immagine così grande che i metodi di correlazione delle immagini richiedono giorni anche con le risorse del supercalcolo.
AIM confronta anche i fotogrammi adiacenti, ma procede ponendo ciascun punto dati al centro di un cerchio (definito dalla precisione della localizzazione) e cercando punti all'interno di quel cerchio in altri fotogrammi. I punti sovrapposti all'interno del "raggio di intersezione" sono condensati in un'unica localizzazione. Quindi il processo viene ripetuto ancora una volta con i punti condensati. Questi passaggi utilizzano risorse computazionali minime e sono più veloci del tempo di acquisizione di una fotocamera per microscopio. Pertanto, è possibile produrre immagini con correzione della deriva in quello che è effettivamente il tempo reale.
I ricercatori hanno testato AIM utilizzando sia dati simulati che strutture chiamate origami di DNA che hanno caratteristiche ben definite. L'algoritmo ha localizzato con successo le strutture e il grado di precisione, inferiore a 1 nanometro, si è rivelato molto più elevato rispetto ai metodi standard di correlazione delle immagini, circa 10 nanometri.
Il laboratorio di Liu incorporerà l'AIM nelle tecniche di microscopia ad alto rendimento sviluppate per migliorare il rilevamento delle malattie. Tuttavia, Liu ritiene anche che l’algoritmo troverà usi in tutta la biologia e la bioingegneria. "È uno strumento veloce e facile da usare e vogliamo renderlo ampiamente accessibile a tutta la comunità", ha affermato. "Stiamo rendendo il nostro software accessibile al pubblico. Vogliamo che le persone ottengano il massimo miglioramento nella risoluzione delle immagini anche solo da questo piccolo intervento di post-elaborazione."
Ulteriori informazioni: Hongqiang Ma et al, Verso la nanoscopia ad alto rendimento senza deriva attraverso la massimizzazione dell'intersezione adattiva, Progressi scientifici (2024). DOI:10.1126/sciadv.adm7765
Informazioni sul giornale: La scienza avanza
Fornito dall'Università dell'Illinois Grainger College of Engineering