Un team di scienziati del Genome Institute di Singapore (GIS) e del Bioinformatics Institute (BII) di A*STAR ha sviluppato un nuovo strumento software di intelligenza artificiale chiamato "BANKSY" che riconosce automaticamente i tipi di cellule presenti in un tessuto, come cellule muscolari, cellule nervose e cellule di grasso.
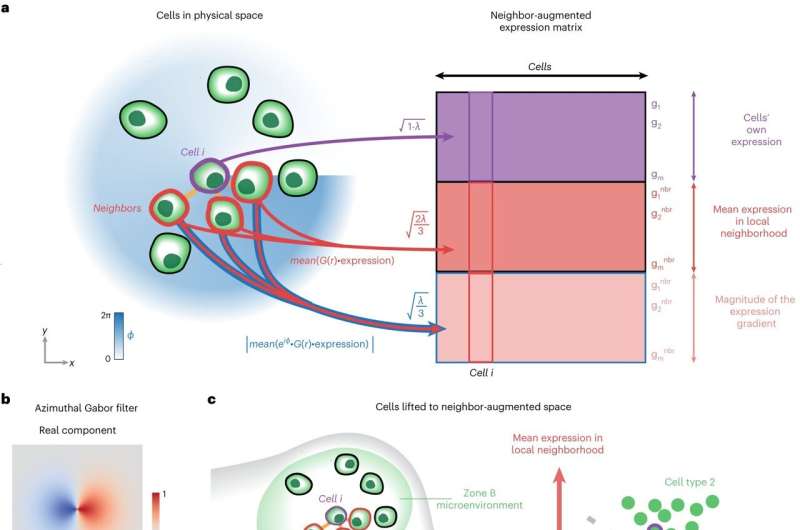
Facendo un passo avanti rispetto agli strumenti convenzionali di intelligenza artificiale, che possono raggruppare le cellule in cluster se contengono molecole simili, BANKSY considera anche quanto siano simili i dintorni delle cellule nel tessuto.
Con BANKSY, i ricercatori sarebbero in grado di migliorare la loro comprensione dei processi tissutali in diverse malattie in modo più rapido e accurato, il che può supportare lo sviluppo di diagnosi e trattamenti più efficaci per il cancro, i disturbi neurologici e altre malattie. Questa ricerca è stata pubblicata nell'articolo "BANKSY unifica la tipizzazione cellulare e la segmentazione dei domini tissutali per l'analisi dei dati omici spaziali scalabili" in Nature Genetics .
BANKSY è abile nell'identificare gruppi cellulari sottilmente distinti nei profili molecolari spaziali generati da campioni di tessuto. Inoltre, BANKSY affronta il problema distinto ma correlato di delimitare regioni anatomiche funzionalmente distinte nelle sezioni di tessuto. Ad esempio, può distinguere strutture stratificate nel prosencefalo umano.
Le tecnologie di profilazione molecolare spaziale (Spatial Omics) sono potenti microscopi che consentono agli scienziati di studiare i tessuti in grande dettaglio rivelando le posizioni esatte delle singole molecole biologiche nelle cellule, nonché la disposizione delle cellule nei tessuti.
Questo li aiuta a capire come le cellule si uniscono nei tessuti per svolgere le loro normali funzioni fisiologiche e anche come si comportano (o si comportano male) in malattie come il cancro, l’autismo o malattie infettive come COVID-19. Questa comprensione è essenziale per una diagnosi più accurata e un trattamento personalizzato dei pazienti, nonché per la scoperta di nuovi farmaci.
BANKSY può aiutare i biologi a interpretare ed estrarre informazioni dalle più recenti tecnologie di Omica Spaziale emerse negli ultimi anni. Versatile, accurato, veloce e scalabile, BANKSY si distingue dai metodi esistenti nell'analisi dei dati Spatial Omics basati su RNA e proteine.
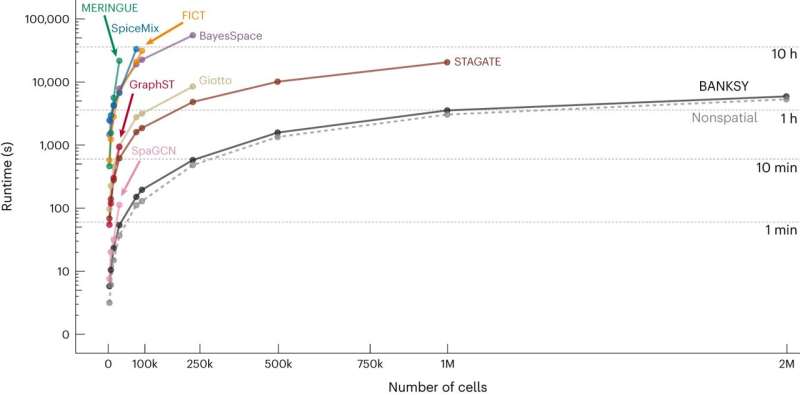
In grado di gestire grandi set di dati di oltre due milioni di celle, BANKSY è da 10 a 1.000 volte più veloce rispetto ai metodi concorrenti testati e da due a 60 volte più scalabile. Ciò significa che il metodo può essere applicato anche ad altre fasi chiave dell'elaborazione dei dati, come il rilevamento e la rimozione di aree di scarsa qualità del campione e l'unione di campioni prelevati da pazienti diversi per un'analisi combinata.
BANKSY è stato valutato in modo indipendente ed è risultato essere l'algoritmo con le migliori prestazioni per i dati di omica spaziale da due studi indipendenti, uno dei quali ha concluso che BANKSY potrebbe essere una potente soluzione per l'identificazione dei domini. L'altro studio ha testato sei algoritmi e ha selezionato BANKSY come il più accurato per l'analisi dei dati.
Il Dr. Shyam Prabhakar, Senior Group Leader presso il Laboratorio di biologia dei sistemi e analisi dei dati e Direttore associato dei sistemi spaziali e unicellulari presso il GIS di A*STAR, ha affermato:"Prevediamo che BANKSY sarà uno strumento rivoluzionario che aiuta a sbloccare il potenziale delle tecnologie emergenti di Omica Spaziale."
"Si spera che ciò migliorerà la nostra comprensione dei processi tissutali in diverse malattie, permettendoci di sviluppare trattamenti più efficaci per tumori, disturbi neurologici e molte altre patologie."
Il professor Liu Jian Jun, direttore esecutivo ad interim del GIS di A*STAR, ha dichiarato:"Il lavoro su BANKSY fa avanzare la nostra strategia di combinare tecnologie ad alto rendimento con software di intelligenza artificiale scalabile e robusto per la risoluzione dei problemi e l'identificazione di indizi su ciò che può fare la differenza nella vita dei pazienti."
Il dottor Iain Tan, consulente senior della Divisione di oncologia medica presso il National Cancer Center di Singapore e scienziato clinico senior presso il laboratorio GIS di genomica applicata del cancro di A*STAR, ha affermato:"Stiamo utilizzando BANKSY per identificare le cellule che aiutano i tumori a crescere e a diffondersi altre parti del corpo:i farmaci mirati a tali cellule potrebbero rappresentare una direzione promettente per il trattamento del cancro."
Ulteriori informazioni: Vipul Singhal et al, BANKSY unifica la tipizzazione cellulare e la segmentazione dei domini tissutali per l'analisi scalabile dei dati omici spaziali, Nature Genetics (2024). DOI:10.1038/s41588-024-01664-3
Informazioni sul giornale: Genetica della natura
Fornito da Agenzia per la scienza, la tecnologia e la ricerca (A*STAR), Singapore