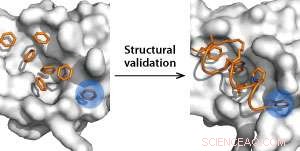
Rilevamento della tasca di legame utilizzando molecole di benzene come sonde (a sinistra) e convalida strutturale utilizzando peptidi graffati (a destra). Credito:Y. S. Tan et al.
Alla ricerca di nuove terapie contro il cancro, I ricercatori di A*STAR hanno ideato una strategia computazionale che porta alla luce siti o "tasche" di legame precedentemente sconosciuti sui bersagli dei farmaci.
È probabile che dalla pipeline di sviluppo dei farmaci emergeranno trattamenti contro il cancro più efficaci. La scoperta di farmaci contro il cancro dipende dall'identificazione e dalla caratterizzazione delle tasche di legame nelle proteine bersaglio. Tipicamente, questa valutazione utilizza tecniche computazionali che si basano su strutture proteiche statiche. Però, le proteine hanno una flessibilità intrinseca che provoca una tendenza a cambiare forma al contatto con i farmaci. Alcune tasche vincolanti rimangono non rilevabili a meno che non interagiscano con una sostanza appropriata e, perciò, vengono perse dalle simulazioni convenzionali. Queste tasche nascoste, però, sono solitamente siti idrorepellenti o idrofobici che si aprono solo quando sono presenti sostanze a bassa polarità.
Per affrontare questo, Yaw Sing Tan e Chandra Verma del Bioinformatics Institute hanno sviluppato un metodo basato su sonde chiamato ligand-mapping Molecular Dynamics (LMMD). Hanno usato questa tecnica per cercare tasche di legame nascoste nella proteina bersaglio antitumorale MDM2. Le previsioni risultanti sono state convalidate sperimentalmente da collaboratori di lunga data del laboratorio p53 di A*STAR e dell'Istituto di scienze chimiche e ingegneristiche, nonché da biologi strutturali dell'Università di Newcastle, UK.
Tan spiega che inizialmente aveva progettato questo metodo basato su sonda per un'altra proteina bersaglio e lo ha utilizzato con successo per trovare una tasca nascosta di legame che è rimasta chiusa nelle simulazioni convenzionali. "Abbiamo quindi deciso di applicare questo approccio all'MDM2 per vedere se potevamo scoprire siti di legame precedentemente sconosciuti che potessero aumentare la potenza degli inibitori MDM2 esistenti, " Aggiunge.
Utilizzando molecole di benzene come sonde di rilevamento di tasche idrofobiche, i ricercatori hanno identificato computazionalmente due nuovi siti di legame su MDM2. "Siamo stati entusiasti di vedere che questi siti si trovano molto vicino alla tasca di legame della proteina soppressore del tumore p53, "dice Tan.
Per di più, i ricercatori si aspettano che i siti appena scoperti portino a peptidi graffati più potenti:si tratta di eliche di amminoacidi stabilizzate chimicamente da una catena di idrocarburi che sono recentemente emerse come potenti attivatori di p53. Di conseguenza, hanno creato peptidi graffati da analoghi noti per legare strettamente MDM2 e riattivare p53, e determinato l'affinità di questi peptidi a MDM2. Le loro simulazioni hanno mostrato che i peptidi hanno legato MDM2 più fortemente di p53 nelle tasche e hanno abbinato esperimenti di cristallografia biofisica e a raggi X.
"Questo metodo potrebbe essere utilizzato per interrogare altri bersagli proteici antitumorali per scoprire nuovi siti di legame che potrebbero essere presi di mira per l'inibizione, " afferma Tan. Il team sta ora lavorando per espandere la portata delle sonde LMMD ad altri tipi di ligandi.