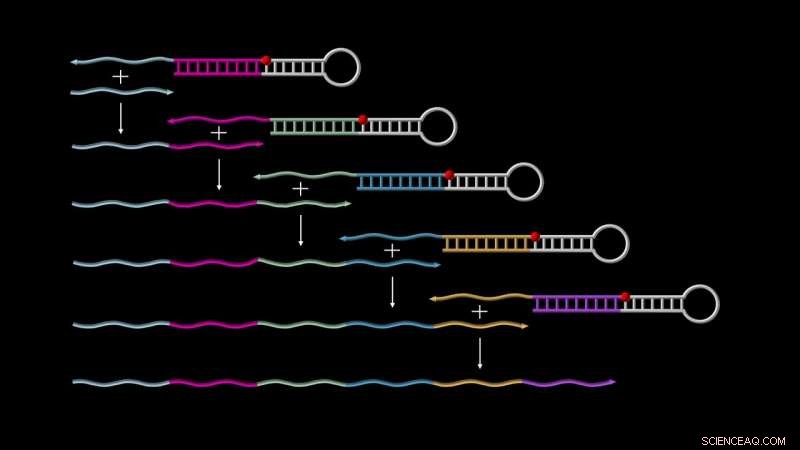
Le cascate di Primer Exchange Reaction (PER) consentono la crescita autonoma di DNA a singolo filamento. Sulla cima, una 'forcina PER catalitica' lega un primo 'primer' (mostrato come un corto filo grigio), innesca il suo allungamento con una sequenza codificata dal tornante stesso, e lo rilascia per iniziare un altro ciclo con il primer già esteso, e così via, fino a quando non viene generata una lunga trascrizione. Credito:Wyss Institute presso l'Università di Harvard
Biologi e nanobiologi sintetici stanno riutilizzando il DNA, il materiale ereditario presente in quasi tutte le cellule del corpo, come materiale autoassemblante intelligente e stabile per costruire nanofabbriche, nanostrutture per la somministrazione di farmaci e dispositivi molecolari in grado di rilevare il loro ambiente e rispondere in modi diversi mediante, Per esempio, rilevare l'infiammazione nel corpo o le tossine nell'ambiente. Queste applicazioni su scala nanometrica spesso implicano la sintesi di grandi sequenze comprendenti migliaia di elementi costitutivi di cui è composto il DNA, noto come A, T, basi nucleotidiche C e G, che può essere ulteriormente ripiegato e strutturato grazie alle specifiche capacità di accoppiamento delle basi tra As e Ts, e C e G, rispettivamente.
Però, finora, i ricercatori non hanno strumenti a loro disposizione che permettano a sequenze a singolo filamento più grandi di crescere autonomamente e quindi unirsi l'una all'altra end-to-end seguendo un piano di progettazione molecolare, una capacità che potrebbe generare strutture e dispositivi con capacità diverse.
Pubblicato oggi in Chimica della natura , la ricerca di Peng Yin presso il Wyss Institute for Biologically Inspired Engineering di Harvard fornisce una soluzione ampiamente applicabile a questo problema. Yin e il suo team hanno sviluppato un metodo che consente a sequenze di DNA pre-progettate di crescere e concatenarsi autonomamente lungo percorsi di assemblaggio specifici, fornendo così la base per una nuova generazione di dispositivi molecolari programmabili. Mettendo alla prova il loro nuovo concetto di cosiddette cascate di "Primer Exchange Reaction" (PER), hanno progettato con successo un primo set di dispositivi con funzioni diverse, come DNA-origami autocostruzione e nanostrutture di DNA che percepiscono, amplificare, registrare o valutare logicamente i segnali ambientali.
I metodi passati producevano copie identiche di una sequenza più piccola fissata, ma non sono in grado di aggiungere sequenze sintetizzate diverse l'una all'altra in modelli definiti per generare in modo autonomo assiemi più grandi senza l'intervento mediato dall'utente. "Le funzionalità autonome e programmabili offerte dalle cascate PER potrebbero generare una generazione completamente nuova di dispositivi e applicazioni molecolari programmabili e colmare le lacune negli sforzi di progettazione, per cui esistono già molte parti mobili, ", ha affermato Peng Yin, membro della facoltà principale del Wyss Institute, dottorato di ricerca, che ha guidato lo studio ed è anche professore di biologia dei sistemi presso la Harvard Medical School (HMS). "Forniamo dati proof-of-concept per PER in una vasta gamma di applicazioni di biologia sintetica all'avanguardia che evidenziano chiaramente l'ampio potenziale della tecnologia".
Il team del Wyss Institute ha utilizzato il nuovo concetto per progettare una serie di tali trascritti PER DNA per applicazioni molto diverse, compresa la sintesi autonoma di grandi nanostrutture di DNA note come DNA-origami, e approcci di biologia sintetica, in cui la sintesi di un trascritto di DNA dipende da un trigger, come un piccolo micro RNA associato al cancro. Il loro approccio PER può persino generare trascrizioni di DNA risultanti da una combinazione valutata logicamente di diversi trigger, simile a RNA Ribocomputing Devices che il team di Yin ha pubblicato all'inizio di quest'anno. interessante, I trascritti PER DNA possono diventare essi stessi catalitici, essere in grado di tagliare un RNA bersaglio arbitrario, diventano sonde fluorescenti che amplificano la presenza di un particolare stimolo molecolare, o "registratori molecolari" che indicano fedelmente l'ordine in cui determinati segnali molecolari appaiono nei loro ambienti.
Per avviare la cascata PER, sono necessari due componenti fondamentali. Uno è chiamato "mediatore a forcina del DNA catalitico", che è una molecola di DNA a singolo filamento che si accoppia parzialmente con se stessa per formare una struttura a forcina con un corto filamento singolo sporgente. Questa sporgenza è progettata per catturare il secondo componente delle cascate PER, il "principio, " che contiene una regione complementare allo sbalzo. Attraverso una serie di reazioni di allungamento e spostamento, il primer viene esteso con una sequenza fornita dal mediatore a forcina catalitica e quindi espulso. Questo libera il mediatore catalitico a forcina per eseguire il ciclo successivo del processo, catturando un nuovo primer di partenza o il primer già allungato, e così via.
Questi complessi percorsi di sintesi procedono in modo autonomo, paragonabile a un robot molecolare che esegue un determinato compito, e ad una sola temperatura, che rende la tecnologia molto robusta. "L'approccio ci offre un'enorme libertà creativa:non solo possiamo sintetizzare lo stesso pezzo di DNA ancora e ancora come nuove aggiunte di una sequenza in crescita, ma possiamo anche variare i tipi di sequenze di DNA da aggiungere semplicemente cambiando la composizione di DNA a forcina catalitica e primer nel mix mentre l'assemblaggio è in corso. Questo ci permette di far diramare la sintesi in direzioni diverse e di modellare in modo complesso la composizione della trascrizione finale del DNA, " ha detto il primo autore dello studio Jocelyn Kishi, che come Graduate Research Fellow della National Science Foundation (NSF) presso HMS lavora nel team del Wyss Institute di Yin. "Stiamo ora lavorando per implementare cascate PER per una varietà di applicazioni, compresi registratori molecolari, diagnostica sofisticata, e imaging tissutale. Speriamo anche che un giorno questi sistemi possano essere utilizzati nelle cellule viventi come dispositivi in grado di registrare eventi o riprogrammare il comportamento cellulare in modi specifici, " disse Kishi.
"Questo nuovo progresso che mostra come le molecole di DNA possono essere programmate per autoassemblarsi in strutture 3D specifiche e svolgere funzioni e compiti predefiniti rappresenta un importante passo avanti nel campo della robotica molecolare, e offre uno sguardo al futuro dei dispositivi per applicazioni mediche e non mediche, ", ha affermato il direttore fondatore del Wyss Institute, Donald Ingber, M.D., dottorato di ricerca, che è anche Judah Folkman Professor of Vascular Biology presso HMS e Vascular Biology Program presso il Boston Children's Hospital, nonché Professore di Bioingegneria presso la Harvard John A. Paulson School of Engineering and Applied Sciences (SEAS).