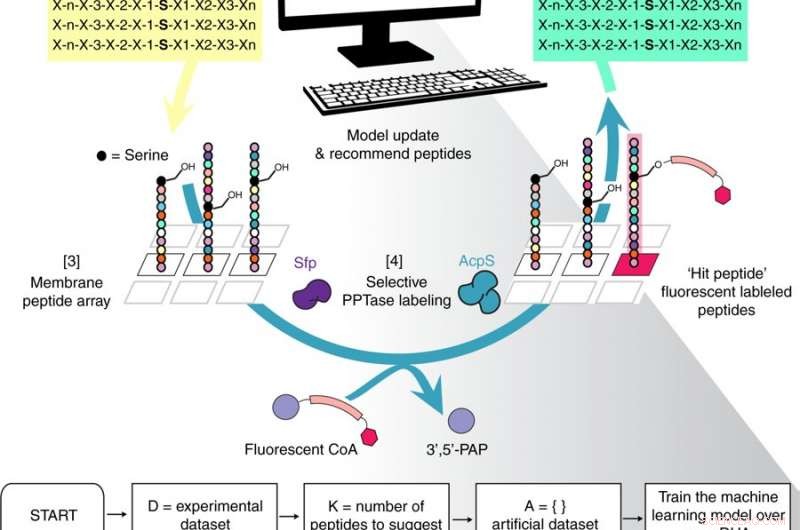
Panoramica del flusso di lavoro iterativo del metodo di ottimizzazione dei peptidi con apprendimento ottimale (POOL). Credito: Comunicazioni sulla natura (2018). DOI:10.1038/s41467-018-07717-6
Scienziati e ingegneri sono da tempo interessati alla sintesi di peptidi, catene di amminoacidi responsabili dello svolgimento di molte funzioni all'interno delle cellule, sia per imitare la natura che per svolgere nuove attività. Un peptide progettato, Per esempio, potrebbe essere un farmaco funzionale che agisce in determinate aree del corpo senza degradarsi, un compito difficile per molti peptidi.
Ma i metodi per scoprire e sintetizzare i peptidi sono costosi e richiedono tempo, spesso implicando mesi o anni di congetture e fallimenti.
ricercatori della Northwestern University, collaborando con i collaboratori della Cornell University e dell'Università della California, San Diego, hanno sviluppato un nuovo modo per trovare sequenze peptidiche ottimali:utilizzare un algoritmo di apprendimento automatico come collaboratore.
L'algoritmo analizza i dati sperimentali e offre suggerimenti sulla successiva migliore sequenza da provare, creando un processo di selezione avanti e indietro che riduce drasticamente il tempo necessario per trovare il peptide ottimale.
I risultati, che potrebbe fornire un nuovo quadro per gli esperimenti nella scienza dei materiali e nella chimica, sono stati pubblicati in Comunicazioni sulla natura il 7 dicembre
"Consideriamo questa come la prossima ondata nel modo in cui progettiamo molecole e materiali, " ha detto il professore della Northwestern Nathan Gianneschi, un autore corrispondente sulla carta. "Possiamo combinare ciò che sappiamo dall'intuizione con la potenza di un algoritmo e trovare la soluzione con meno esperimenti".
Gianneschi è Jacob and Rosaline Cohn Professor nel dipartimento di chimica del Weinberg College of Arts and Sciences della Northwestern e nei dipartimenti di scienza e ingegneria dei materiali e di ingegneria biomedica presso la Northwestern Engineering.
Per creare il metodo, Gianneschi, che è anche direttore associato dell'International Institute for Nanotechnology della Northwestern, ha collaborato con Peter Frazier, un professore associato alla Cornell che lavora nella ricerca operativa e nell'apprendimento automatico, e Michael Burkart, un biologo chimico ed esperto in enzimologia presso l'UC San Diego, per trovare un modo migliore per creare peptidi che potrebbero generare biomateriali, in particolare nanostrutture e microstrutture che potrebbero modificare le proteine in determinati modi. Il primo passo è stato trovare i peptidi giusti che avrebbero agito come substrati enzimatici per queste strutture.
I peptidi sono costituiti da catene di amminoacidi che possono essere lunghe fino a 20 amminoacidi, con 20 diverse possibilità per ogni acido. Poiché la sequenza determina la funzione del peptide, la determinazione delle sequenze ottimali richiede costosi esperimenti, spesso condotti con congetture.
Gli sperimentalisti, Gianneschi e Burkart, ha lavorato con Frazier per diversi anni per sviluppare un sistema che combinasse i dati sperimentali con un algoritmo di apprendimento automatico per trovare le migliori strategie per la creazione di nuovi materiali.
Dopo che Frazier ha progettato l'algoritmo e i due hanno lavorato insieme per addestrarlo, gli sperimentali hanno sviluppato una serie di 100 peptidi, ha condotto esperimenti per capire quali funzionavano come avrebbero dovuto, quindi immesso tali informazioni nell'algoritmo. L'algoritmo ha quindi consigliato cosa cambiare per il prossimo ciclo di sviluppo del peptide, e ha anche raccomandato strategie che pensava fallissero.
"Ora stavamo iniziando a ottenere selettività, " ha detto Gianneschi. Completando questo processo più volte, sono stati in grado di raggiungere i peptidi ottimali.
"Invece di indovinare e guardare milioni di peptidi, siamo stati in grado di guardare centinaia di peptidi e convergere molto rapidamente su sequenze che si sono comportate in modi completamente nuovi, " ha detto. Se confrontato con mutazioni casuali o congetture, il metodo dell'algoritmo ha avuto statisticamente molto più successo.
Sebbene questo lavoro si sia concentrato sui substrati, questo processo potrebbe essere utilizzato per scoprire peptidi per qualsiasi tipo di scopo, come la consegna della droga, e forse anche essere usato per scoprire sequenze di DNA, anche. Poiché si potrebbe scoprire qualsiasi tipo di sequenza ottimale, i ricercatori inoltre non si limitano a quali sequenze di aminoacidi si trovano nel codice genetico.
Il prossimo passo sarà automatizzare l'intero processo. Gianneschi è anche interessato ad utilizzare il metodo per trovare superfici ottimali per i polimeri, specificamente polimeri utilizzati negli impianti medici. Trovare le superfici giuste che si legano ai tessuti o ai muscoli potrebbe aiutare a prevenire il tessuto cicatriziale o il rigetto dell'impianto.
"Potresti essenzialmente scoprire sequenze che fanno cose specifiche, che è davvero al centro di ciò che i peptidi e gli acidi nucleici fanno in natura, " ha detto. "Questo potrebbe rivoluzionare il modo in cui produciamo i peptidi".