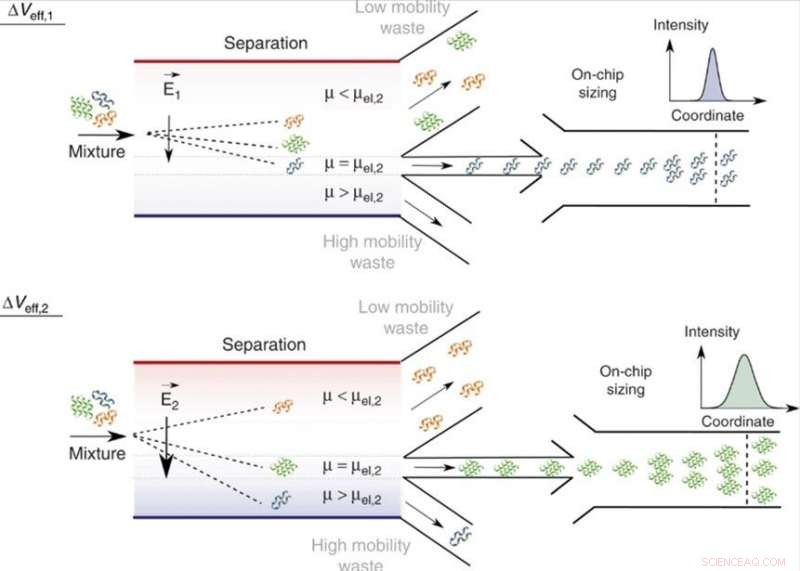
Separazione su chip e analisi di miscele in fase liquida. Regolando la tensione effettiva, Veff, molecole di una specifica mobilità elettroforetica, μ = μel, e quindi di una carica specifica al rapporto raggio idrodinamico, qRh, possono essere indirizzati all'area di analisi con quelli di minore (μ < μel) e maggiore (μ > μel) di mobilità diretti ai canali di raccolta dei rifiuti. Poiché la tensione applicata può essere variata, è necessaria una sola unità di analisi e la larghezza della camera di separazione può essere mantenuta limitata, consentendo al dispositivo di mantenere un'elevata efficienza di tensione e una velocità di elaborazione rapida. Nell'area di analisi, le frazioni vengono dimensionate tramite dimensionamento diffusionale microfluidico (MDS) monitorando il loro movimento spazio-temporale in condizioni di flusso laminare. Credito:Microsistemi e nanoingegneria, doi:10.1038/s41378-019-0072-3
I sistemi microfluidici sono utilizzati in biologia molecolare, biochimica e biotecnologia per analizzare rapidamente miscele biomolecolari eterogenee con elevate velocità di recupero e volumi minimi di campione. Però, è difficile combinare processi preparativi e analitici all'interno di un singolo dispositivo per un'analisi rapida e integrata. In un recente studio ora pubblicato su Microsistemi e Nanoingegneria , Kadi L. Saar e collaboratori presso i dipartimenti interdisciplinari di chimica, fisica, e Fluidic Analytics Limited a Cambridge, UK., hanno sviluppato un chip che combina le due fasi di preparazione e analisi.
Inizialmente, hanno usato la tensione per separare le molecole proteiche all'interno di una miscela binaria di biomolecole di uguali dimensioni indistinguibili tramite tecniche di dimensionamento o risoluzione convenzionali. Successivamente, il team di ricerca ha utilizzato il nuovo dispositivo per ottenere un'impronta digitale 2D di una miscela proteica eterogenea. I risultati apriranno nuove possibilità per acquisire dati multiparametrici rapidi su sistemi biomolecolari in tempi brevi.
Le tecniche di microfluidica sono interessanti per l'analisi di campioni biologici a causa dei requisiti di campione molto bassi e dell'elevato tasso di recupero. Le piattaforme possono fornire una velocità di analisi insuperabile a livello di singole unità operative o fornire più unità con flussi di lavoro direttamente combinati, senza trasferimento del campione tra le unità. Tali trasferimenti avvengono attraverso connettori o tubi e introducono dispersione nel campione, che influenzano le prestazioni del sistema. Il flusso di lavoro qui proposto può separare miscele eterogenee per determinare i componenti di interesse e ridurre le complessità per l'ulteriore elaborazione della miscela per la sua purificazione.
I ricercatori avevano precedentemente introdotto una varietà di strategie di separazione molecolare basate sul flusso continuo su scala micron, compresa l'elettroforesi a flusso libero, dielettroforesi, magnetoforesi e separazione acustoforetica. Strategie di rilevamento come fluorescenza indotta da laser o LED (LIF), chemiluminescenza o approcci elettrochimici possono essere radicati in parallelo all'interno di tali piattaforme di separazione microfluidica. Le informazioni analitiche sui composti separati possono essere ottenute con strategie offline come la spettroscopia di massa o SDS-PAGE, ma le tecniche possono limitare la velocità di elaborazione in un singolo dispositivo, causando la perdita o la contaminazione del campione.
Saar et al. ha quindi sviluppato una separazione completamente integrata e una caratterizzazione quantitativa di campioni biomolecolari eterogenei in un singolo dispositivo microfluidico per superare i limiti esistenti accoppiando direttamente la separazione su chip all'analisi su chip e al dimensionamento molecolare. La caratteristica del progetto ha permesso l'analisi di una frazione specifica regolando l'intensità di campo applicata. Hanno progettato il dispositivo per identificare le frazioni separate simili ai metodi SEC-MALS (cromatografia ad esclusione dimensionale con diffusione della luce multiangolo) o LC (chip)-MS ((su chip)-cromatografia liquida-spettrometria di massa).
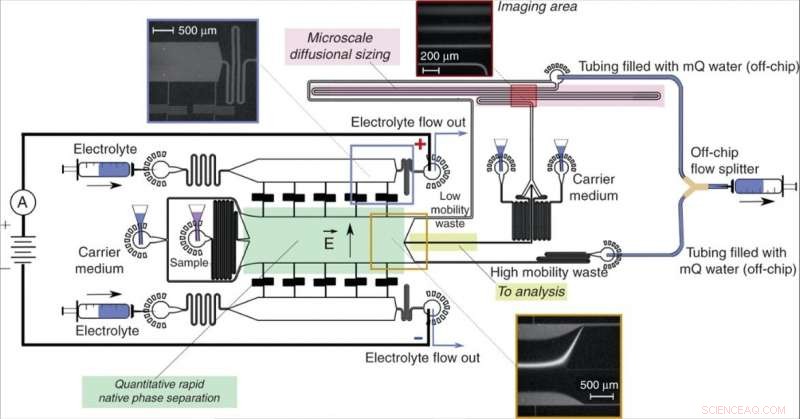
Progettazione e funzionamento del dispositivo. Un'unità di elettroforesi a flusso libero (verde), consentendo una rapida separazione delle molecole di analita nella loro fase nativa e fornendo informazioni quantitative sul processo di separazione, frazioni dirette del campione (giallo) a un processo di analisi a valle che coinvolge il dimensionamento diffusionale microfluidico (MDS; rosa). Il campione è stato caratterizzato monitorando un singolo frame di imaging (riquadro in alto a destra) per estrarre contemporaneamente le dimensioni e le cariche delle frazioni separate. Il dispositivo è stato azionato applicando una pressione negativa alla sua uscita con un divisore di flusso off-chip a forma di Y mantenendo separate le soluzioni dai canali "rifiuti a bassa mobilità" e "rifiuti ad alta mobilità" per evitare cortocircuiti parziali del dispositivo. Il potenziale elettrico è stato applicato dalle uscite della soluzione elettrolitica impiegando una soluzione elettrolitica fluente come descritto in precedenza13. Il flusso della soluzione elettrolitica, che includeva un tracciante fluorescente per la sua visualizzazione, è stato scelto in modo tale che l'elettrolita raggiungesse la sua uscita anziché essere prelevato nella camera di separazione senza raggiungerlo (riquadro in alto a sinistra), eppure si sarebbe infiltrato nella camera a distanza controllata, lasciando spazio sufficiente per deviare il raggio del campione (riquadro inferiore). Credito:Microsistemi e nanoingegneria, doi:10.1038/s41378-019-0072-3
Il dispositivo ha avuto l'ulteriore vantaggio di eseguire l'intero processo completamente radicato per la separazione elettroforetica in soluzione libera, consentendo ai ricercatori di ottenere una mappa quantitativa in pochi minuti, molto più velocemente delle tecniche convenzionali. La miscela non è stata influenzata dal mezzo di supporto, ei ricercatori potrebbero studiare sia le interazioni molecolari deboli che quelle non covalenti.
Saar et al. ha progettato il dispositivo utilizzando un'unità di elettroforesi quantitativa in fase nativa collegata a un'unità di dimensionamento del dispositivo di diffusione microfluidica (MDS). La piattaforma combinata ha consentito componenti di mobilità elettroforetica specifica (µ e1 ) per l'analisi a valle su chip, in funzione dell'intensità del campo elettrico applicato. Hanno progettato tre canali a valle dell'unità di elettroforesi per mantenere i prodotti dell'elettrolisi lontano dal chip senza entrare nel dispositivo. Hanno ridotto al minimo il numero di singole unità che hanno guidato il flusso nel dispositivo, accoppiato al funzionamento stabile del dispositivo per la caratterizzazione quantitativa del campione. Gli scienziati hanno tenuto le uscite dell'elettrolita separate dal dispositivo combinato per applicare il potenziale elettrico attraverso il dispositivo senza generare un cortocircuito elettrico, e per consentire la rimozione efficiente di qualsiasi prodotto dell'elettrolisi senza accumulo per evitare fluttuazioni di pressione.
Il team di ricerca ha applicato il potenziale elettrico su connettori metallici per generare un'interfaccia metallo-fluido all'esterno del chip in conformità con il prototipo del dispositivo progettato dallo stesso team. In questo lavoro, Saar et al. ha progettato un divisore di flusso a forma di Y e ha mantenuto i flussi separati fino a quando non hanno raggiunto il divisore per evitare cortocircuiti parziali. Hanno calcolato la portata dell'elettrolita nel dispositivo per avere un effetto sulle prestazioni del dispositivo.
(a) La tensione applicata attraverso la camera di elettroforesi è stata regolata in passaggi lineari per indirizzare specifiche frazioni all'analisi. (b) Le frazioni sono state quindi dimensionate mediante imaging dell'unità di dimensionamento diffusionale costituita da quattro canali in cui è stata monitorata l'entità della diffusione delle molecole di analita nel tampone di supporto circostante. (c) La dimensione media delle molecole di analiti in ciascuna delle frazioni è stata trovata adattando i profili fluorescenti osservati per diversi raggi idrodinamici (Rh) e minimizzando l'errore quadratico medio minimo tra l'adattamento (linea tratteggiata rossa) e i dati (linea tratteggiata rossa) linea continua). (d) L'intensità della fluorescenza nell'area di analisi varia a seconda della concentrazione degli analiti in ciascuna delle frazioni (in alto) e le dimensioni dei componenti in una miscela binaria di lisozima e albumina di siero bovino sono risultate essere Rh = 1.9 nm e Rh = 3.6 nm (in basso). (e) Questa miscela binaria non può essere caratterizzata utilizzando tecniche di dimensionamento che non implichino passaggi di prefrazionamento, come la diffusione dinamica della luce (DLS; linea tratteggiata azzurra). Il suo dimensionamento è stato possibile utilizzando il dispositivo descritto in questo lavoro (linea continua blu scuro). Credito:Microsistemi e nanoingegneria, doi:10.1038/s41378-019-0072-3 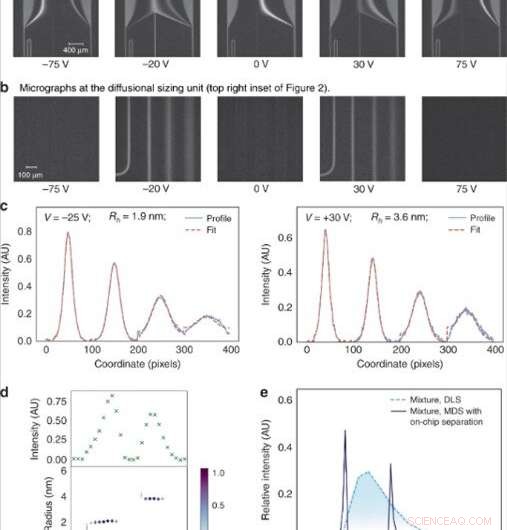
Hanno ripreso l'ugello in corrispondenza del quale il campione ha incontrato il mezzo di trasporto come punto di riferimento sul movimento delle particelle. Gli scienziati hanno regolato il canale di dimensionamento della diffusione o la portata per dimensionare con precisione le molecole di analita, ordini di grandezza maggiori o minori. Poiché hanno progettato la piattaforma di microfluidica utilizzando Poly (dimethylsiloxane) (PDMS), gli scienziati hanno eliminato qualsiasi autofluorescenza nella configurazione prima di analizzare i dati dell'immagine.
Hanno quindi utilizzato il dispositivo per analizzare una miscela binaria di proteine campione; albumina di siero bovino e lisozima umano. Per preservare gli stati nativi delle molecole proteiche, hanno ripreso i campioni privi di etichetta con un microscopio a lunghezza d'onda UV costruito in casa e quantificato la fluorescenza intrinseca del campione. Saar et al. ha confermato la capacità di separare la miscela nei suoi componenti applicando prima una serie di tensioni per registrare i profili fluorescenti. Hanno quindi registrato la mobilità elettroforetica delle proteine (µ e1 ) combinato con la portata nel dispositivo per caratterizzare la maggior parte delle proteine e dei loro complessi. Gli scienziati hanno alterato la portata o la tensione applicata per analizzare le biomolecole con diversi parametri biofisici.
Utilizzando la piattaforma, hanno caratterizzato rapidamente miscele di molecole su scala nanometrica, dove i singoli analiti hanno mostrato dimensioni simili ma diverse proprietà elettroforetiche. Sulla base dell'istogramma risultante, il gruppo di ricerca ha confermato la presenza di due campioni distinti. Comparativamente, in un approccio di separazione convenzionale off-chip, quest'ultimo passaggio richiedeva il frazionamento mediante trasferimento del campione da uno strumento analitico a un altro tramite tubi interconnessi, limitando le prestazioni del dispositivo. La concentrazione proteica totale nello studio si è avvicinata a 100 µM e gli scienziati hanno rilevato con precisione il limite di sensibilità a circa 100 nM, rispetto alla fluorescenza intrinseca delle frazioni proteiche. Per composti otticamente non attivi, Saar et al. suggerire una strategia alternativa di rilevamento e caratterizzazione come il rilevamento della massa secca.
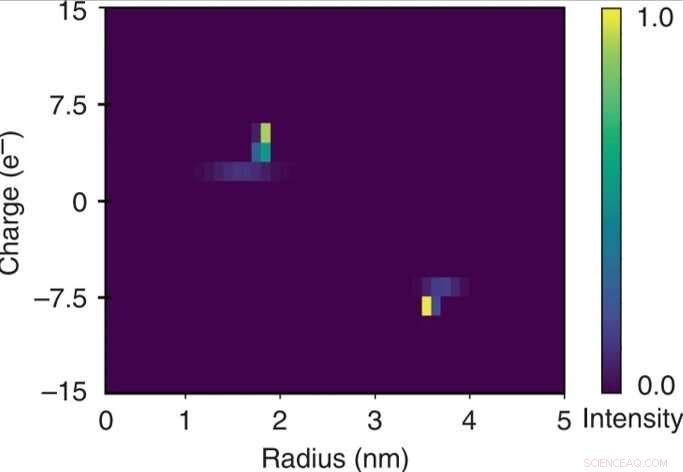
Caratterizzazione bidimensionale rapida di una miscela di albumina sierica bovina e lisozima umano. La natura quantitativa dell'unità di separazione elettroforetica ha permesso di mettere in relazione il potenziale applicato con la mobilità elettroforetica μel di ciascuna delle frazioni. Questi dati sono stati utilizzati in combinazione con i raggi idrodinamici estratti (Rh) per stimare le cariche effettive (q) delle molecole in ciascuna delle frazioni e per costruire una mappa bidimensionale q vs Rh della miscela su un arco temporale di pochi minuti da monitorare il dispositivo solo a un singolo frame di imaging. Credito:Microsistemi e nanoingegneria, doi:10.1038/s41378-019-0072-3
Saar et al. ha utilizzato la strategia per ottenere mappe caratteristiche bidimensionali (2-D) della miscela proteica come prova di concetto. Hanno estratto informazioni quantitative dalla fase di separazione e hanno collegato i potenziali applicati alle mobilità elettroforetiche della specie per stimare l'efficacia del dispositivo. Hanno registrato la corrente che scorre nel sistema durante il normale funzionamento e quando la camera di separazione è stata cortocircuitata per stimare la resistenza elettrica totale del dispositivo e degli elettrodi.
I ricercatori hanno calcolato la mobilità elettroforetica come il movimento di una particella in un campo elettrico per ciascuna delle frazioni. Sulla base dei dati sperimentali, la mappa caratteristica 2-D costruita includeva la carica effettiva (q) e il raggio idrodinamico (Rh) della miscela. Le unità di carica elementari risultanti delle proteine specifiche concordavano con i valori stimati altrove. Hanno ottenuto la mappa bidimensionale completa monitorando solo un singolo frame di imaging per un'analisi rapida della soluzione.
Il tempo analitico del dispositivo di microfluidica dalla separazione al dimensionamento diffusionale e all'imaging è di circa 14 secondi. Gli scienziati hanno costruito la mappa sperimentale 2-D utilizzando solo 3 µl di campione in sette minuti in totale, ordini di grandezza più veloci rispetto alla scala temporale per eseguire gel proteici 2-D convenzionali. Il team di ricerca ha condotto un'ampia gamma di interazioni biomolecolari, in soluzione, direttamente in condizioni native che in precedenza erano difficili da eseguire in laboratorio.
In questo modo, Kadi L. Saar e collaboratori hanno sviluppato un dispositivo microfluidico che combina la separazione su chip con l'analisi diretta su chip per sostituire gli approcci convenzionali su microscala esistenti. Utilizzando il dispositivo, hanno rapidamente analizzato una miscela binaria di proteine che non è stato possibile identificare come componenti individuali tramite gli approcci di dimensionamento delle soluzioni esistenti. Hanno costruito una mappa caratteristica 2-D della miscela eterogenea su una scala temporale rapida per aprire la possibilità di caratterizzazione delle proteine in soluzione a una risoluzione temporale senza precedenti rispetto alle tecniche biofisiche esistenti.
© 2019 Scienza X Rete