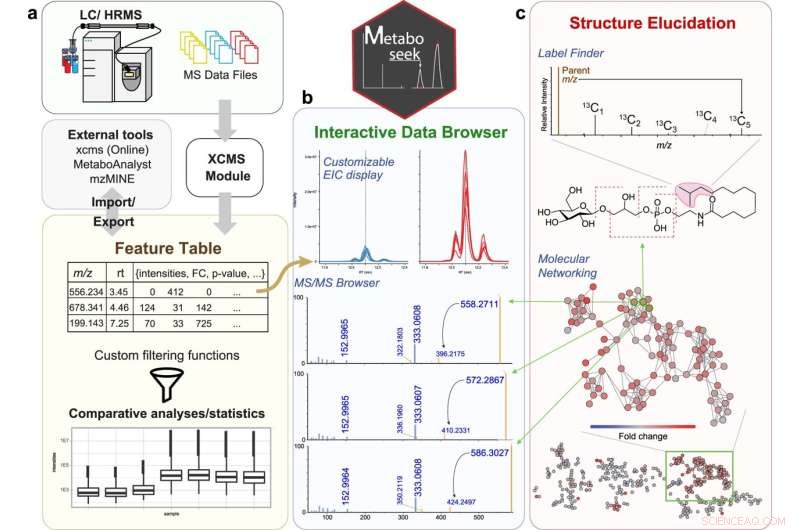
Metabolomica comparativa con Metaboseek. a Metaboseek include un modulo XCMS integrato per il rilevamento delle funzionalità e il raggruppamento delle funzionalità (con annotazione CAMERA) e accetta tabelle delle funzionalità generate da altri software. b Le funzioni possono essere annotate e ordinate per priorità utilizzando ampie opzioni di filtraggio e strumenti statistici integrati. È possibile sfogliare rapidamente i dati grezzi per ciascuna caratteristica molecolare, inclusi gli spettri EIC, MS1 e MS/MS associati. c Il browser dei dati interagisce con una suite di strumenti di delucidazione della struttura, ad esempio formula molecolare basata su SIRIUS e previsione della struttura, Label Finder per identificare i composti marcati con isotopi e il cercatore di pattern MS/MS per identificare le caratteristiche della SM con pattern di frammentazione caratteristici. Credito:Comunicazioni sulla natura (2022). DOI:10.1038/s41467-022-28391-9
In qualità di ricercatore post-dottorato nel laboratorio del membro della facoltà BTI Frank Schroeder, Max Helf ha visto i suoi compagni di laboratorio lottare continuamente durante l'analisi dei dati. Quindi, ha deciso di fare qualcosa al riguardo e ha sviluppato un'app open source gratuita chiamata Metaboseek, che ora è essenziale per il lavoro del laboratorio.
Il laboratorio Schroeder studia il nematode Caenorhabditis elegans, uno dei sistemi modello di maggior successo per la biologia umana, per scoprire nuovi metaboliti che governano le vie di segnalazione evolutivamente conservate e potrebbero essere utili come cavi per lo sviluppo di nuovi prodotti farmaceutici o agrochimici. I ricercatori svolgono questo compito confrontando i metaboliti tra due diverse popolazioni di vermi, un processo chiamato metabolomica comparativa.
Dato che i campioni contengono normalmente più di 100.000 composti, gli approcci computazionali sono essenziali per eseguire l'analisi.
Il team si era affidato a pacchetti software che non offrivano il livello di flessibilità richiesto per personalizzare facilmente i parametri di analisi. Quella limitazione, e la mancanza di un'interfaccia utente grafica adeguata, significava che i colleghi di Helf hanno dovuto affrontare il compito ingombrante di ispezionare visivamente cumuli di dati, ad esempio per individuare possibili falsi positivi, e saltare tra diversi altri strumenti software per confermare e filtrare quelli privi di significato risultati.
"Mi è sembrato molto inefficiente e non sono riuscito a superare le carenze di altre soluzioni software per questo problema", ha affermato Helf. "Pensavo che ci dovesse essere un modo più semplice, così ho iniziato a scrivere codice per il mio software."
Helf ha sviluppato la versione iniziale del suo software nel 2017 e ha continuato a migliorarla nei due anni successivi. "Oltre ad affrontare i problemi che i miei compagni di laboratorio stavano già affrontando, ho parlato con loro di cos'altro li tratteneva - cosa volevano fare ma non stavano nemmeno provando - e ho creato quelle funzionalità nell'app", ha detto Helf, che ora è un product manager di bioinformatica presso l'azienda di proteomica Biognosys AG. "Volevo che questo nuovo strumento fosse facile da usare e accessibile a chiunque si occupi di biologia chimica."
Il risultato è stato Metaboseek, un'app con un'interfaccia grafica che incorpora molteplici strumenti di analisi dei dati che i ricercatori non codificanti altrimenti non avrebbero. L'app semplifica l'analisi dei dati metabolomici comparativi aiutando il ricercatore a determinare quali caratteristiche dei dati sono reali e consentendo loro di approfondire tali caratteristiche, il tutto all'interno dello stesso strumento.
"Max l'ha fatto senza che me lo chiedessi", ha detto Schroeder. "Prima che sapessi che stava succedendo, c'era Metaboseek. Abbiamo iniziato a usarlo e ora il nostro laboratorio e molti collaboratori non potrebbero esistere senza di esso."
In uno studio pubblicato su Nature Communications il 10 febbraio, il team di Schroeder ha fornito la prova del concetto per Metaboseek applicandolo a un importante percorso del metabolismo dei grassi che non era ancora stato studiato:il percorso di α-ossidazione in C. elegans che aiuta ad abbattere una classe di acidi grassi.
Utilizzando Metaboseek, il team ha scoperto che i nematodi privi di un gene chiave nel percorso di α-ossidazione accumulavano centinaia di metaboliti non segnalati in precedenza. I risultati sono importanti perché l'α-ossidazione è una via biochimica di base nei vermi che si conserva negli esseri umani, ha detto Schroeder.
"Bennett Fox ha svolto il lavoro di chimica, quindi questo studio è stata una bella collaborazione tra i due dottorandi", ha aggiunto Schroeder, che è anche professore al Dipartimento di Chimica e Biologia Chimica della Cornell University.
Secondo Schroeder e Helf, ci sono alcuni motivi per cui non ci sono molti buoni strumenti analitici per confrontare i dati metabolomici. In primo luogo, la metabolomica comparativa è un campo relativamente giovane rispetto ad altri campi della biologia ricchi di dati come la genomica (che si concentra sul DNA) e la proteomica (che si concentra sulle proteine), quindi non c'è stato abbastanza tempo per sviluppare strumenti software e infrastrutture di database .
Inoltre, nell'ultimo decennio, l'avvento di spettrometri di massa convenienti e ad altissima risoluzione per la raccolta di dati metabolomici ha aumentato forse di oltre dieci volte la quantità di dati che un campione può generare, creando un'esigenza ancora maggiore di strumenti sofisticati in grado di mantenere al passo con la marea di dati.
Metaboseek soddisfa queste esigenze con una serie di funzionalità per l'analisi di vari tipi di dati per aiutare l'identificazione dei composti, la determinazione della struttura, l'assegnazione dei metaboliti alle famiglie in base a somiglianze strutturali, il tracciamento dei composti radiomarcati e altro ancora. + Esplora ulteriormente