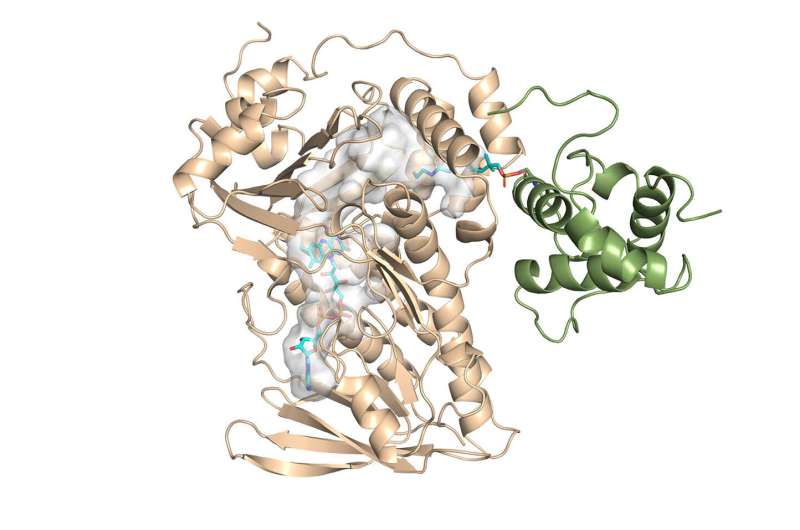
I chimici hanno determinato per la prima volta la struttura cristallina di un componente chiave dell'enzima monensina e hanno svelato il meccanismo della sua attività di reazione.
"La scoperta principale è stata la prima struttura cristallina di questa famiglia di enzimi", ha affermato Chu-Young Kim, professore di biochimica presso l'Università dell'Illinois Urbana-Champaign (UIUC), che ha guidato la parte sperimentale dello studio. Lui e i suoi colleghi hanno risolto la struttura cristallina del MonCI, un enzima chiave nei batteri del suolo che sintetizza naturalmente la monensina.
Lela Vukovic, professoressa associata presso l'Università del Texas, a El Paso (UTEP), ha eseguito gli studi computazionali sulla ricerca sul monensina, pubblicati su Nature Communications .
L’iniziativa dell’Università del Texas Research Cyberinfrastructure (UTRC) ha assegnato allocazioni di supercomputer Vukovic sul sistema Lonestar6 presso il Texas Advanced Computing Center (TACC) per affrontare queste sfide. UTRC fornisce funzionalità informatiche avanzate ai ricercatori di tutte le 14 istituzioni del sistema UT.
Lonestar6 del TACC ha contribuito a rivelare la sequenza di reazione che produce monensina. Questa ricerca apre la strada alla progettazione futura di antibiotici più sicuri ed efficaci.
"Abbiamo scoperto che MonCI viene utilizzato per effettuare tre reazioni cruciali di epossidazione", ha detto Kim. "Questo è molto insolito e ha implicazioni su come possiamo ingegnerizzare il batterio per produrre nuovi antibiotici."
Kim, che ha recentemente lasciato l'UTEP per unirsi all'UIUC, ha consultato il laboratorio di Vukovic con i risultati della struttura e pieno di nuove domande. Ciò che ha scoperto è stata un'interessante reazione sequenziale all'interno dell'enzima. Tuttavia, era ancora sperimentalmente impossibile ottenere la struttura cristallina dell'enzima con il substrato all'interno nella sua modalità attiva.
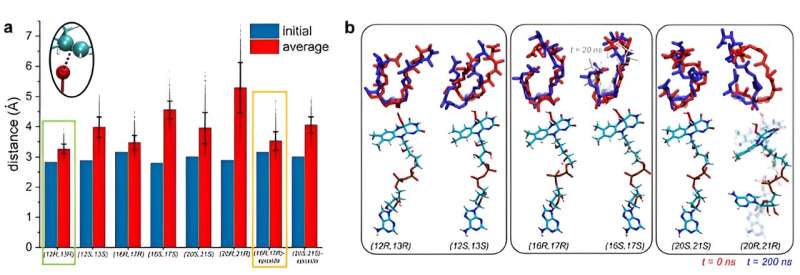
Fu allora che Kim e Vukovic decisero di modellare l'enzima e il sottostato in simulazioni in cui il substrato è stabile. Se un substrato è stabile in una determinata posizione, può verificarsi una reazione per quella posizione.
Vukovic e i suoi studenti Tara Nitka e Anju Yadav hanno sviluppato modelli completi del sistema di circa 78.000 atomi basati sulle strutture cristalline determinate dagli sperimentali dello studio.
"Le sfide computazionali sono nate dall'esame di più sistemi per determinare la posizione in cui la premonensina A e le sue versioni epossidate sono più stabili e hanno maggiori probabilità di subire la prima, la seconda e la terza reazione di epossidazione", ha affermato Vukovic.
"I supercomputer sono stati molto utili nel caratterizzare queste molecole biologiche che producono naturalmente antibiotici", ha aggiunto Vukovic. "Non saremmo in grado di eseguire studi computazionali senza di essi. Gli studi computazionali ci aiutano a scoprire e comprendere queste complesse reazioni sequenziali importanti per la società."
Vukovic ha svolto la sua ricerca post-dottorato presso l'UIUC sotto la guida del compianto Klaus Schulten, la cui eredità sopravvive nel software NAMD sviluppato dal suo gruppo e utilizzato in questo studio e in innumerevoli altri.
"L'UIUC sta lavorando molto per ottimizzare il NAMD affinché possa essere eseguito sui supercomputer nazionali, come Lonertar6 e Stampede2 al TACC", ha affermato. "NAMD ci ha permesso di approfondire questo enzima e vedere quali reazioni avvengono per prima, seconda e terza per generare monensina."
"La biosintesi della monensina richiede almeno 14 enzimi diversi, uno dei quali è MonCI", ha aggiunto Kim. "Dobbiamo anche studiare tutti gli altri enzimi. In futuro, prevediamo di generare versioni migliorate del monensina per prenderci cura meglio del bestiame e del pollame. Inoltre, il monensina è tossico per cavalli e cani, quindi questi animali da fattoria vengono talvolta avvelenati accidentalmente e ucciso. Pertanto è necessario un monensin non tossico."
Gli autori sono Qian Wang, Tara A. Nitka, Anju Yadav e Lela Vukovic dell'Università del Texas a El Paso; Ning Liu, Hongli Xiao, Hui Yang e Xi Chen della Northwest University, Cina; Irimpan I. Mathews dello SLAC National Accelerator Laboratory; e Chu-Young Kim dell'Università dell'Illinois Urbana-Champaign.
Ulteriori informazioni: Qian Wang et al, Formazione di triepossido da parte di una monoossigenasi flavina-dipendente nella biosintesi della monensina, Nature Communications (2023). DOI:10.1038/s41467-023-41889-0
Fornito dall'Università del Texas ad Austin