Un team di Amherst dell'Università del Massachusetts ha fatto un grande passo avanti verso la modellizzazione e la comprensione del modo in cui le proteine intrinsecamente disordinate (IDP) subiscono la separazione di fase spontanea, un importante meccanismo di organizzazione subcellulare che è alla base di numerose funzioni biologiche e malattie umane.
Gli sfollati interni svolgono un ruolo cruciale nel cancro, nei disturbi neurodegenerativi e nelle malattie infettive. Costituiscono circa un terzo delle proteine prodotte dal corpo umano e due terzi delle proteine associate al cancro contengono segmenti o domini grandi e disordinati. Identificare le caratteristiche nascoste cruciali per il funzionamento e l'auto-assemblaggio degli sfollati interni aiuterà i ricercatori a capire cosa non va in queste caratteristiche quando si verificano le malattie.
In un articolo pubblicato sul Journal of American Chemical Society , l'autore senior Jianhan Chen, professore di chimica, descrive un nuovo modo per simulare le separazioni di fase mediate dagli IDP, un processo importante che è stato difficile da studiare e descrivere.
"La separazione di fase è un fenomeno molto noto nella fisica dei polimeri, ma ciò che la gente non sapeva fino a circa 15 anni fa era che questo è un fenomeno molto comune anche in biologia", spiega Chen. "Si può osservare la separazione di fase al microscopio, ma comprendere questo fenomeno a livello molecolare è molto difficile.
"Negli ultimi cinque o dieci anni, le persone hanno iniziato a scoprire che molte di queste proteine disordinate possono guidare la separazione di fase, comprese numerose importanti proteine coinvolte nel cancro e nei disturbi neurodegenerativi."
Il nuovo articolo, basato sulla ricerca nel laboratorio di biofisica computazionale e biomateriali di Chen, costituisce un capitolo del dottorato di ricerca dell'autore principale Yumeng Zhang. tesi. Zhang inizierà a lavorare come ricercatore post-dottorato presso il Massachusetts Institute of Technology (MIT) a febbraio. Un altro collaboratore chiave è Shanlong Li, un ricercatore associato post-dottorato nel laboratorio di Chen.
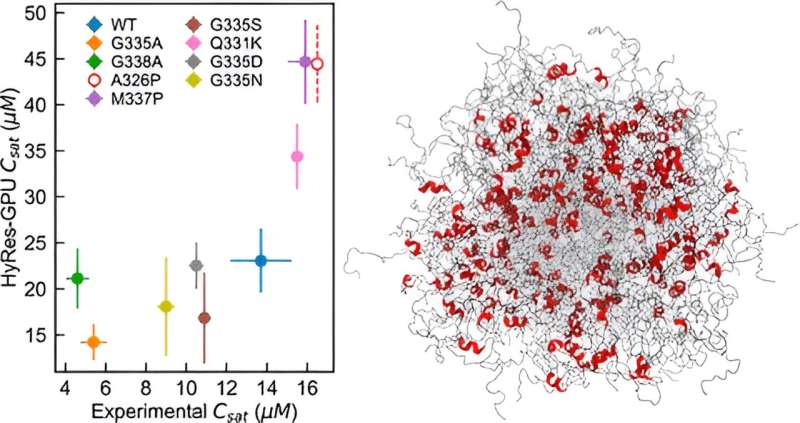
Il laboratorio di Chen ha sviluppato un campo di forza accurato con risoluzione ibrida (HyRes) accelerata dalla GPU per simulare le separazioni di fase mediate dagli IDP. Questo modello è unico nella sua capacità di descrivere accuratamente le interazioni della struttura peptidica e le strutture secondarie transitorie, pur essendo sufficientemente efficiente dal punto di vista computazionale per modellare la separazione di fase liquido-liquido. Questo nuovo modello colma una lacuna critica nella capacità esistente nella simulazione al computer della separazione di fase IDP.
Chen e il suo team hanno creato simulazioni HyRes per dimostrare per la prima volta cosa governa la stabilità del condensato di due importanti IDP.
"In realtà non avevo previsto che potesse fare un lavoro così buono nel descrivere la separazione di fase perché è un fenomeno davvero difficile da simulare", dice Chen. "Abbiamo dimostrato che questo modello è sufficientemente accurato per iniziare a osservare gli impatti anche di una singola mutazione o di strutture residue nella separazione di fase."
HyRes-GPU dei ricercatori fornisce uno strumento di simulazione innovativo per studiare i meccanismi molecolari della separazione di fase. L'obiettivo finale è sviluppare strategie terapeutiche nel trattamento delle malattie associate a proteine disordinate.
"Questo è davvero il significato di questo lavoro", afferma Chen. "Si ritiene che importanti processi biologici avvengano attraverso la separazione di fase. Quindi, se riusciamo a capire meglio cosa controlla questo processo, quella conoscenza sarà davvero potente, se non essenziale, per pensare al controllo della separazione di fase per vari scopi scientifici e ingegneristici. Questo ci aiuterà a capire il tipo di intervento che sarà necessario per ottenere effetti terapeutici."
Chen afferma che il prossimo passo sarà applicare ciò che il suo team ha imparato a simulazioni su scala più ampia di miscele biomolecolari più complesse.
"Shanlong sta ora lavorando alla costruzione di un modello simile per gli acidi nucleici perché la separazione di fase spesso coinvolge sia proteine disordinate che acidi nucleici", afferma. "Vogliamo essere in grado di descrivere entrambi i componenti chiave e questo ci consentirebbe di esaminare molti più sistemi."
Ulteriori informazioni: Yumeng Zhang et al, Verso una simulazione accurata dell'accoppiamento tra la struttura secondaria delle proteine e la separazione di fase, Journal of the American Chemical Society (2023). DOI:10.1021/jacs.3c09195
Informazioni sul giornale: Giornale dell'American Chemical Society
Fornito dall'Università del Massachusetts Amherst