Utilizzando Cas13 strutturato, i ricercatori della Rice University e dell'Università del Connecticut hanno modificato uno strumento di editing genetico per fungere da test diagnostico altamente sensibile per la presenza del virus SARS-CoV-2. Hanno utilizzato un sensore elettrochimico standard per fornire risultati. Credito:Jie Yang/Rice University
Un metodo ingegnerizzato basato su CRISPR che trova l'RNA da SARS-CoV-2, il virus che causa COVID-19, promette di rendere i test per questa e altre malattie facili e veloci.
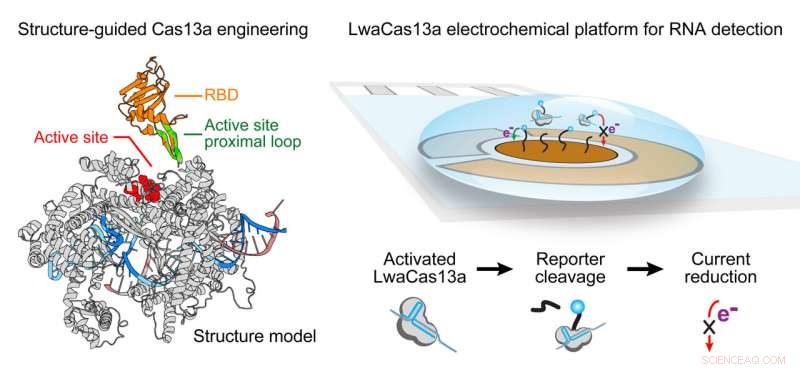
I collaboratori della Rice University e dell'Università del Connecticut hanno ulteriormente ingegnerizzato il sistema CRISPR-Cas13 di modifica dell'RNA per aumentare la loro potenza per rilevare quantità minime del virus SARS-CoV-2 in campioni biologici senza la fase di estrazione e amplificazione dell'RNA che richiede tempo in test PCR gold standard.
La nuova piattaforma ha avuto molto successo rispetto alla PCR, trovando 10 positivi su 11 e nessun falso positivo per il virus nei test su campioni clinici direttamente da tamponi nasali. I ricercatori hanno mostrato che la loro tecnica trova segni di SARS-CoV-2 nell'attomolare (10 -18 ) concentrazioni.
Lo studio condotto dall'ingegnere chimico e biomolecolare Xue Sherry Gao della George R. Brown School of Engineering della Rice e dai ricercatori post-dottorato Jie Yang della Rice e Yang Song del Connecticut appare in Nature Chemical Biology .
Cas13, come il suo cugino più noto Cas9, fa parte del sistema attraverso il quale i batteri si difendono naturalmente dai fagi invasori. Dalla sua scoperta, CRISPR-Cas9 è stato adattato dagli scienziati per modificare i genomi del DNA vivente e mostra grandi promesse per il trattamento e persino la cura delle malattie.

Il ricercatore post-dottorato della Rice University Jie Yang ha condotto uno sforzo per adattare gli strumenti di modifica del genoma Cas13 per fungere da rivelatore altamente sensibile per la presenza del virus SARS-CoV-2, che causa COVID-19. Credito:Jeff Fitlow/Rice University
E può essere utilizzato in altri modi. Cas13 da solo può essere potenziato con l'RNA guida per trovare e intercettare sequenze di RNA bersaglio, ma anche per trovare "collaterali", in questo caso la presenza di virus come SARS-CoV-2.
"La proteina Cas13 ingegnerizzata in questo lavoro può essere prontamente adattata ad altre piattaforme precedentemente stabilite", ha detto Gao. "La stabilità e la robustezza delle varianti Cas13 ingegnerizzate le rendono più adatte per la diagnostica point-of-care in aree con risorse limitate quando non sono disponibili costose macchine PCR."
Yang ha detto che il Cas13 wild-type, estratto da un batterio, Leptotrichia wadei, non è in grado di rilevare il livello attomolare di RNA virale in un intervallo di tempo compreso tra 30 e 60 minuti, ma la versione migliorata creata alla Rice fa il lavoro in circa mezz'ora e rileva la SARS -CoV-2 in concentrazioni molto inferiori rispetto ai test precedenti.
Ha detto che la chiave è un anello flessibile ben nascosto vicino al sito attivo di Cas13. "È nel mezzo della proteina vicino al sito catalitico che determina l'attività di Cas13", ha detto Yang. "Dato che Cas13 è grande e dinamico, è stato difficile trovare un sito per inserire un altro dominio funzionale".
I ricercatori hanno fuso sette diversi domini di legame dell'RNA al ciclo e due dei complessi erano chiaramente superiori. Quando trovavano i loro bersagli, le proteine diventavano fluorescenti, rivelando la presenza del virus.
"Abbiamo potuto vedere che l'aumento dell'attività era cinque o sei volte superiore a Cas13 di tipo selvaggio", ha detto Yang. "Questo numero sembra piccolo, ma è piuttosto sorprendente con un singolo passaggio di ingegneria delle proteine.
"Ma ciò non era ancora sufficiente per il rilevamento, quindi abbiamo spostato l'intero test da un lettore di lastre a fluorescenza, che è abbastanza grande e non disponibile in impostazioni a basse risorse, a un sensore elettrochimico, che ha una sensibilità maggiore e può essere utilizzato per punti -of-care diagnostics," she said.
With the off-the-shelf sensor, Yang said the engineered protein was five orders of magnitude more sensitive in detecting the virus compared to the wild-type protein.
The lab wants to adapt its technology to paper strips like those in home COVID-19 antibody tests, but with much higher sensitivity and accuracy. "We hope that will make testing more convenient and with lower cost for many targets," Gao said.
The researchers are also investigating improved detection of the Zika, dengue and Ebola viruses and predictive biomarkers for cardiovascular disease. Their work could lead to rapid diagnosis of the severity of COVID-19.
"Different viruses have different sequences," Yang said. "We can design guide RNA to target a specific sequence that we can then detect, which is the power of the CRISPR-Cas13 system."
But because the project began just as the pandemic took hold, SARS-CoV-2 was a natural focus. "The technology is quite amenable to all the targets," she said. "This makes it a very good option to detect all kinds of mutations or different coronaviruses."
"We are very excited about this work as a combinational effort of structure biology, protein engineering and biomedical device development," Gao added. "I greatly appreciate all the efforts from my lab members and collaborators."
Co-authors of the paper are Rice postdoctoral researcher Xiangyu Deng, undergraduate Jeffrey Vanegas and graduate student Zheng You; graduate students Yuxuan Zhang and Zhengyan Weng of the University of Connecticut; microbiology supervisor Lori Avery and Kevin Dieckhaus, a professor of medicine, of UConn Health; Yi Zhang, an assistant professor of biomedical engineering at the University of Connecticut; and Yang Gao, an assistant professor of biosciences at Rice. + Esplora ulteriormente