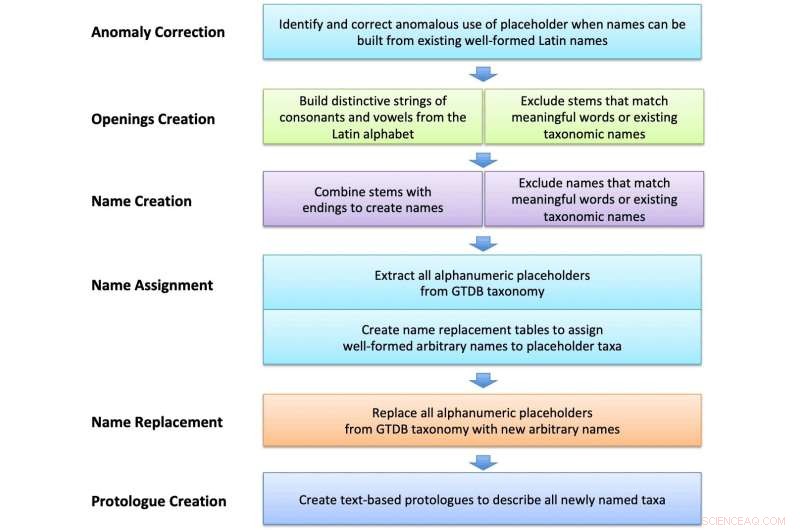
Flusso di lavoro schematico per la creazione e l'assegnazione di nomi latini arbitrari. Le stringhe costruite con lettere latine sono state selezionate e curate per escludere componenti indesiderati e parole significative. Questi sono stati combinati con suffissi o parole latine per creare nomi, che sono stati quindi utilizzati per nominare taxa segnaposto nei file GTDB e creare protologhi basati su testo. Credito:Giornale internazionale di microbiologia sistematica ed evolutiva (2022). DOI:10.1099/ijsem.0.005482
In un articolo recentemente pubblicato sull'International Journal of Systematic and Evolutionary Microbiology , ricercatori nel Regno Unito e in Austria hanno nominato più di 65.000 diversi tipi di microbi. Lo studio, guidato dal professor Mark Pallen al Quadram Institute di Norwich, attinge a una lunga tradizione di creazione di nomi latini ben formati ma arbitrari per nuove specie, ma applica questo approccio su una scala senza precedenti nella storia della tassonomia.
Il Prof. Pallen sostiene che la microbiologia è una vittima del suo stesso successo, con decine di migliaia di nuove specie scoperte negli ultimi anni, ma la maggior parte rimane senza nome. In passato, ai batteri sono stati dati nomi descrittivi o hanno preso il nome da persone o luoghi. Questo approccio sta attualmente fornendo circa mille nuovi nomi di specie all'anno. Tuttavia, con un arretrato di oltre 50.000 specie ben classificate ma senza nome, a questo ritmo di progresso, ci vorrebbe almeno mezzo secolo per nominare tutti questi batteri senza nome, a quel punto gli scienziati dovrebbero affrontare il problema di nominare milioni di specie in più scoperto nel frattempo.
Pallen e colleghi hanno adottato un approccio efficiente, ad alto rendimento e basato sui big data, utilizzando un programma per computer per generare decine di migliaia di nomi distintivi ma facili da usare che non hanno alcuna somiglianza con le parole esistenti. Per soddisfare il requisito che i nomi dei batteri debbano essere in latino, il team ha combinato stringhe arbitrarie di lettere dell'alfabeto latino con suffissi femminili grammaticalmente ben formati. Il risultato è un insieme di nomi che richiamano la familiarità e la gravità del latino, anche se privi di un pedigree significativo. Gli esempi includono Dupisella tifacia per un batterio dell'intestino delle pecore, Hopelia gocarosa per un batterio che vive nelle acque sotterranee svedesi o Saxicetta apufaria da un lago salato russo.
Sebbene ciò possa sembrare radicale, in realtà la formazione dei nomi in modo arbitrario ha una lunga tradizione, che risale al primo codice di nomenclatura tassonomica nel 1869 e addirittura al padre della tassonomia, il naturalista svedese Linneo. Ciò che è diverso qui è lo stupefacente senso della scala, con un catalogo di nomi che corre per oltre diecimila pagine in quella che rappresenta la più grande denominazione di specie mai vista in una singola pubblicazione.
Poiché sono stati applicati a specie scoperte dal sequenziamento del DNA ma non ancora coltivate in laboratorio, per ora i nuovi nomi rimangono provvisori piuttosto permanenti. Tuttavia, poiché il codice della nomenclatura insiste sul fatto che i batteriologi mirano alla stabilità dei nomi e non sono in corso approcci concorrenti di denominazione su scala, sembra probabile che la stragrande maggioranza dei nuovi nomi verrà utilizzata per anni o addirittura secoli a venire.
In chiusura, Pallen afferma:"Sono stato un membro del gruppo di lavoro che ci ha fornito lettere greche per le varianti COVID, che sono state rapidamente adottate dalla comunità scientifica. Spero che anche i nomi qui proposti siano adottati rapidamente e ampiamente utilizzati. Questo è solo il primo passo. L'era della scoperta microbica è tutt'altro che finita, ma sarà facile creare nomi futuri in massa usando i principi che abbiamo stabilito qui". + Esplora ulteriormente