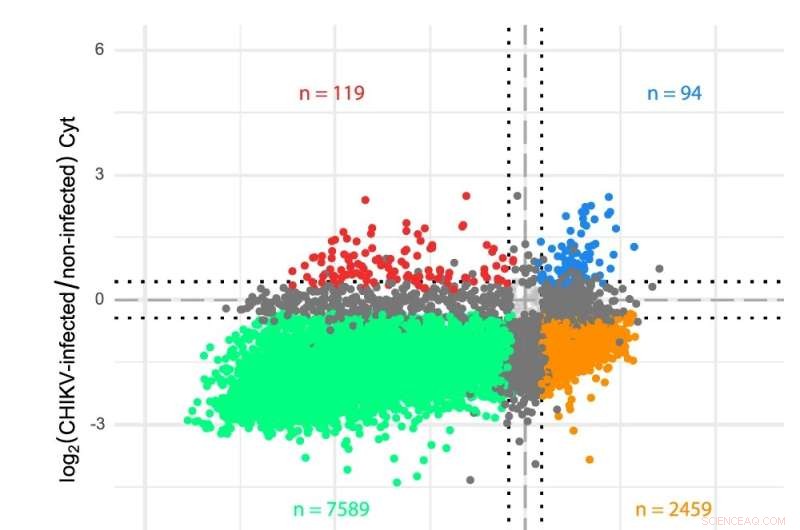
uno schema del genoma dell'RNA CHIKV. b Analisi del trascrittoma del citosol (Cyt) e dei compartimenti ER in cellule infettate da CHIKV. I geni sono stati tracciati in base ai loro cambiamenti di piega log2 nelle cellule infette da CHIKV rispetto a quelle non infette per entrambi i compartimenti di traduzione. Le letture del sequenziamento sono state analizzate utilizzando un'annotazione del trascrittoma personalizzata e l'espressione differenziale è stata determinata tramite limma-voom (vedi Metodi). I colori indicano geni che sono significativamente (FDR < 0,05) down-regolati in entrambi i compartimenti (verde), up-regolati in entrambi i compartimenti (blu), up-o down-regolati in un solo compartimento (grigio scuro) o mostrano un comportamento opposto in i due scomparti (arancione e rosso). Le linee tratteggiate indicano variazioni log2 volte di ± 0,433 (cioè una variazione del 35% in entrambe le direzioni) utilizzate come soglia minima per dichiarare i livelli di mRNA come modificati. Le cellule HEK 293T sono state infettate a un MOI di 4 e i campioni sono stati raccolti a dodici ore dopo l'infezione. Per ciascuna condizione sono state effettuate tre repliche indipendenti. Credito:Comunicazioni sulla natura DOI:10.1038/s41467-022-31835-x
Il Molecular Virology Research Group dell'Università Pompeu Fabra (UPF), in collaborazione con il gruppo Epitranscriptomics and RNA Dynamics del Center for Genomic Regulation (CRG), ha scoperto un nuovo meccanismo per cui i virus modificano il macchinario cellulare per leggere meglio le istruzioni nel genoma del virus invasore e quindi produrre elevate quantità di progenie virale. Lo studio è stato pubblicato su Nature Communications ed è stato guidato da Juana Díez.
I geni contengono le informazioni necessarie per la formazione delle proteine, molecole complesse essenziali per la vita, formate da amminoacidi. La lettura di queste informazioni avviene in due fasi principali, la prima è la trascrizione, in cui l'informazione del gene (DNA) viene trasferita a una molecola chiamata RNA messaggero (mRNA). L'mRNA è costituito da un "testo" formato da triplette di nucleotidi (le lettere GCT, CAT, ecc.). Ogni tripletta corrisponde a un amminoacido. La seconda fase è la traduzione, in cui una molecola chiamata RNA di trasferimento (tRNA) riconosce ciascuna tripletta e funge da traduttore portando l'amminoacido corrispondente. Le proteine vengono costruite tramite questo processo.
Ci sono 61 codoni e 20 amminoacidi, e così tante triplette codificano per lo stesso amminoacido. Ogni organismo utilizza preferibilmente una di queste triplette (tripletta ottimale) perché ha una concentrazione maggiore del tRNA che riconosce quella tripletta. Pertanto, quando il "testo" dell'mRNA è arricchito in triplette ottimali, le proteine saranno generate in modo rapido ed efficiente mentre quando sono arricchite in triplette non ottimali, l'efficienza dell'espressione diminuirà perché i relativi tRNA sono scarsi.
I virus sono molto semplici e per moltiplicarsi ed esprimere le loro proteine hanno bisogno di dirottare il macchinario cellulare dell'ospite. I virus generano il proprio mRNA nelle cellule che infettano, che queste ultime leggono e generano proteine virali per produrre più virus. Ma gli mRNA di molti virus, tra cui SARS-CoV-2 e virus trasmessi dalle zanzare dengue, zika e chikungunya, sono arricchiti in triplette non ottimali ed esprimono ancora proteine virali con grande efficacia. "Per affrontare questo dilemma, abbiamo utilizzato il virus chikungunya come modello perché il suo genoma si moltiplica a livelli estremamente elevati", spiegano Jennifer Jungfleisch e René Böetcher, coautori dello studio.
"I nostri risultati mostrano per la prima volta che i virus modificano il tRNA dell'ospite per adattare il meccanismo di traduzione dell'ospite al testo dell'mRNA virale", afferma Marc Talló, anche coautore dell'articolo. "In altre parole, l'infezione virale induce un cambiamento del linguaggio nella cellula, in modo che esprima le proteine virali in modo molto efficiente. Poiché le proteine virali sono essenziali per la produzione di virus, in definitiva questo cambiamento sarà responsabile della generazione di un numero elevato di virus nella cellula infetta", aggiunge.
"Sebbene lo studio si sia concentrato sul virus chikungunya, la nostra proposta è che la modifica dei tRNA indotta dall'infezione virale sia un meccanismo generale seguito da molti virus", spiega Juana Díez, professore ordinario presso il Dipartimento di Medicina e Scienze della Vita dell'UPF.
"Inoltre, i nostri risultati forniscono una base per considerare la regolazione del tRNA come un nuovo e promettente obiettivo terapeutico per lo sviluppo di antivirali ad ampio spettro che sono efficaci contro più virus", conclude Díez. Lo studio ha coinvolto anche il gruppo di ricerca coordinato da Eva María Novoa al CRG, e gli altri autori sono Gemma Pérez-Vilaró e Andres Merits (Istituto di Tecnologia, Università di Tartu). + Esplora ulteriormente