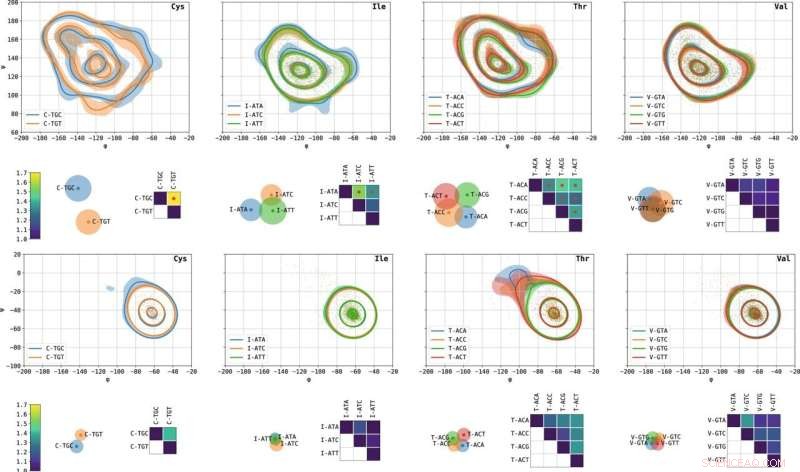
Grafici Ramachandran specifici del codone di amminoacidi selezionati e distanze tra di loro. Da sinistra a destra sono mostrate cisteina, isoleucina, treonina e valina. I grafici di contorno rappresentano le linee di livello contenenti il 10, 50 e 90% della massa di probabilità. Le regioni ombreggiate rappresentano intervalli di confidenza del 10%–90% calcolati su 1000 bootstrap casuali. Sono rappresentate le modalità β- (in alto) e α- (in basso). Le matrici mostrano L1 distanze tra coppie di grafici Ramachandran specifici del codone, normalizzati in modo che l'autodistanza sia 1. I punti rossi indicano coppie con distribuzioni dell'angolo diedro significativamente diverse in base al loro valore p. I grafici a dispersione che visualizzano le matrici di distanza sono stati ottenuti da una variante del ridimensionamento multidimensionale (MDS). Ogni punto rappresenta un codone; le distanze euclidee a coppie tra i punti approssimano L1 distanza tra i codoni corrispondenti. I cerchi approssimano i raggi di incertezza. Più due cerchi si sovrappongono, meno distinguibili sono i corrispondenti grafici Ramachandran specifici del codone. Credito:Comunicazioni sulla natura (2022). DOI:10.1038/s41467-022-30390-9
Uno studio che integra idee biologiche e nuovi strumenti informatici ha scoperto nuove associazioni tra codificazione genetica e struttura delle proteine, che potrebbero potenzialmente cambiare il modo in cui pensiamo alla produzione di proteine nel ribosoma, la "catena di assemblaggio delle proteine" della cellula. La ricerca, guidata dal professor Alex Bronstein, dalla dottoressa Ailie Marx e dal dottorato di ricerca. studente Aviv Rosenberg, è stato pubblicato su Nature Communications .
Le proteine, le molecole complesse che svolgono ruoli critici praticamente in ogni meccanismo biologico, sono prodotte dai ribosomi in un processo chiamato traduzione. Il ribosoma decodifica le "istruzioni genetiche" in arrivo per sintetizzare catene di aminoacidi, i mattoni delle proteine. Quando gli amminoacidi sono legati in sequenza in una lunga catena, si ripiegano in una struttura tridimensionale unica che garantisce alla proteina le sue proprietà biologiche e la sua funzionalità. Errori di traduzione possono portare a ripiegamenti errati e successivamente a disturbi fisiologici, sia lievi che gravi.
Le istruzioni per la produzione di proteine vengono consegnate al ribosoma come codoni, sequenze di tre "lettere" dal codice nucleotidico genetico, che specificano l'identità e l'ordine degli amminoacidi che devono essere aggiunti dal ribosoma alla catena proteica. Ad esempio, il codone UUU segnala l'aggiunta dell'amminoacido fenilalanina, mentre il codone UAC indica l'aggiunta di tirosina. In questo modo, la sequenza del codone codifica per la sequenza unica di amminoacidi caratteristica di ciascuna proteina. Questa mappatura dei codoni genetici sugli amminoacidi utilizzati nella traduzione è comune a tutte le creature viventi del pianeta ed è considerata un meccanismo primordiale.
Come se tutto ciò non fosse abbastanza complicato, è importante sottolineare che ci sono 61 codoni che vengono decodificati in soli 20 aminoacidi. In altre parole, tutti gli amminoacidi tranne due sono codificati da più codoni.
È qui che entra in gioco la presente ricerca. Sulla base di esperimenti condotti negli anni '60 e '70, il dogma accettato afferma che le proteine non portano "memoria" del codone specifico da cui ciascun amminoacido è stato tradotto fintanto che l'identità dell'amminoacido rimane invariata. Questi primi esperimenti sul ripiegamento delle proteine utilizzavano denaturanti chimici per dispiegare proteine completamente formate e poi hanno dimostrato che dopo la rimozione di queste sostanze chimiche la catena proteica poteva ripiegarsi spontaneamente per riguadagnare la sua struttura e funzione originali. Questi esperimenti hanno suggerito che solo la sequenza di amminoacidi, e non la sequenza specifica del codone, determina la struttura di una proteina. Alla luce di questo dogma, le mutazioni che modificano il codice genetico senza modificare l'amminoacido sono ampiamente definite "silenziose" e considerate irrilevanti per la struttura e la funzione delle proteine.
Il team di ricerca Technion ha scoperto un'associazione tra l'identità del codone e la struttura locale della proteina tradotta, il che suggerisce che questo potrebbe non essere il caso generale e che le proteine potrebbero effettivamente "ricordare" le istruzioni specifiche da cui sono state sintetizzate. Il team di ricerca ha analizzato migliaia di strutture proteiche tridimensionali utilizzando strumenti dedicati da loro sviluppati, che integrano metodi informatici avanzati, apprendimento automatico e statistica. In questo modo, hanno confrontato accuratamente le distribuzioni degli angoli formati in queste strutture sotto diversi codici genetici sinonimi. I loro risultati mostrano che per alcuni codoni esiste una significativa dipendenza statistica tra l'identità del codone e la struttura locale della proteina, nella posizione dell'amminoacido codificato da quel codone.
I ricercatori sottolineano che i risultati non sono ancora in grado di far luce sulla direzione della relazione causale, il che significa che non è ancora possibile dire se un cambiamento nel codice genetico possa causare un cambiamento nella struttura proteica locale o se cambiamenti strutturali possano causare codificazione diversa, ad esempio attraverso processi evolutivi. This question is the foundation for a subsequent research study now being carried out by the group. According to Dr. Marx, a biologist by training and education, "If we find in subsequent research that the codon indeed has a causal effect on protein folding, this is likely to have a huge impact on our understanding of protein folding, as well as on future applications, such as engineering new proteins."
Dr. Marx emphasizes that the discovery presented in the article would not have been possible without Prof. Bronstein's computer and analysis skills. "This research is truly interdisciplinary, because biology alone cannot cope with such vast quantities of data without the help of data science, and computer scientists cannot themselves perform research of this kind, since they lack familiarity with the complex biological processes being probed. Therefore, our research highlights the huge advantage of interdisciplinary research that integrates skills from different fields to create a whole that is greater than the sum of its parts."