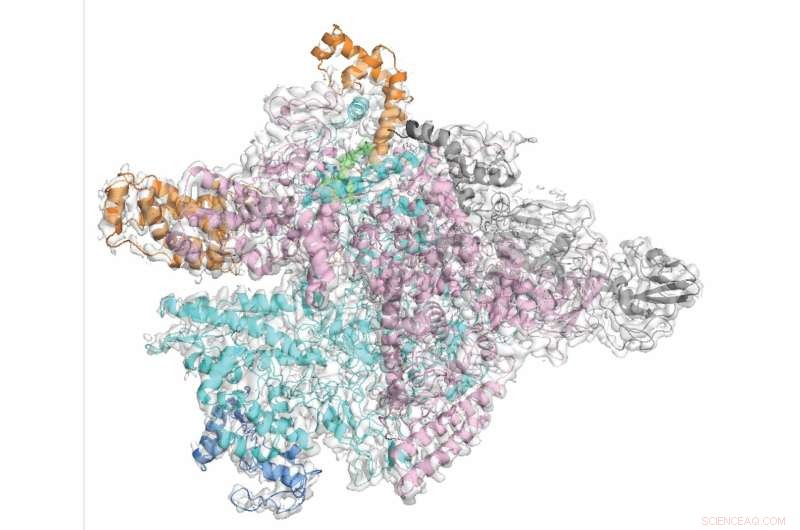
I ricercatori hanno dimostrato a livello molecolare come la fidaxomicina prende di mira selettivamente C. diff risparmiando gli innocenti astanti batterici. Credito:The Rockefeller University
La maggior parte degli antibiotici sono spade a doppio taglio. Oltre a uccidere l'agente patogeno per cui sono prescritti, decimano anche i batteri benefici e cambiano la composizione del microbioma intestinale. Di conseguenza, i pazienti diventano più inclini alla reinfezione ed è più probabile che emergano ceppi resistenti ai farmaci.
La risposta a questo problema potrebbe essere rappresentata dagli antibiotici a spettro ristretto che uccidono solo una o poche specie di batteri, riducendo al minimo il rischio di danni collaterali. In uno studio recente, gli scienziati di Rockefeller hanno esaminato da vicino uno di questi antibiotici, la fidaxomicina, usato per trattare il Clostridium difficile, o C. diff, una delle infezioni più comuni associate all'assistenza sanitaria. I ricercatori hanno dimostrato a livello molecolare come la fidaxomicina prende di mira selettivamente C. diff risparmiando gli innocenti astanti batterici.
I risultati, dettagliati in Natura , potrebbe aiutare gli scienziati nella corsa allo sviluppo di nuovi antibiotici a spettro ristretto contro altri agenti patogeni.
"Voglio che le persone, gli scienziati e i medici pensino in modo diverso agli antibiotici", afferma Elizabeth Campbell, professore associato di ricerca al Rockefeller. "Poiché il nostro microbioma è fondamentale per la salute, gli approcci a spettro ristretto hanno un ruolo importante da svolgere nel modo in cui tratteremo le infezioni batteriche in futuro."
Enigmaticamente selettivo
C. diff è un batterio produttore di tossine che può infiammare il colon e causare grave diarrea. Infetta circa mezzo milione di persone negli Stati Uniti, per lo più in un ambiente ospedaliero, e circa uno su 11 di quelli di età superiore ai 65 anni che muore entro un mese.
Per anni, i medici hanno utilizzato antibiotici ad ampio spettro per il trattamento di C. diff. La fidaxomicina è un'alternativa relativamente nuova che ha ottenuto l'approvazione della FDA nel 2011.
Come molti altri antibiotici, incluso il farmaco per la tubercolosi rifampicina, la fidaxomicina prende di mira un enzima chiamato RNA polimerasi (RNAP), che il batterio usa per trascrivere il suo codice DNA in RNA. Per capire esattamente perché la fidaxomicina inibisce selettivamente RNAP in C. diff e non nella maggior parte degli altri batteri, Campbell ha collaborato con il biochimico Robert Landick dell'Università del Wisconsin-Madison per visualizzare C. diff RNAP utilizzando la microscopia crioelettronica, una potente tecnica di imaging che può rivelare la forma 3D delle molecole e catturare la molecola del farmaco e il suo bersaglio in azione. "Sebbene l'architettura complessiva di RNAP in diversi batteri sia simile, ci sono ancora differenze considerevoli", afferma Campbell.
Spiare RNAP
Una grande sfida, tuttavia, è stata quella di produrre prima grandi quantità di C. diff, un germe anaerobico che non cresce in presenza di ossigeno. Il primo autore dello studio, Xinyun Cao, del Landick Lab, ha trascorso due anni a sviluppare un sistema per produrre più facilmente C. diff RNAP utilizzando E. Coli, un batterio di facile crescita utilizzato frequentemente in laboratorio.
Usando questo materiale, il co-primo autore Hande Boyaci, un post-dottorato del team di Campbell, ha generato immagini di C. diff RNAP bloccato con fidaxomicina a una risoluzione quasi atomica. Incastrati in un cardine tra due subunità di RNAP, gli inceppamenti di fidaxomicina aprono la pinza dell'enzima, impedendogli di aggrapparsi al materiale genetico e avviare il processo di trascrizione.
Nell'esaminare da vicino i punti di contatto tra RNAP e fidaxomicina, i ricercatori hanno identificato un aminoacido sull'RNP che si lega alla fidaxomicina ma è assente nei principali gruppi di microbi intestinali risparmiati dalla fidaxomicina. Una versione geneticamente modificata di C. diff che mancava di questo aminoacido era imperturbabile dalla fidaxomicina, proprio come altri batteri commensali nell'intestino. Al contrario, i batteri che lo avevano aggiunto al loro RNAP sono diventati sensibili alla fidaxomicina.
I risultati suggeriscono che questo amminoacido tra i 4.000 aminoacidi di questa macchina di trascrizione robusta ed essenziale sia il suo tallone d'Achille, responsabile dell'uccisione dei batteri da parte della fidaxomicina.
L'approccio utilizzato in questo studio propone una tabella di marcia per lo sviluppo di antibiotici nuovi e più sicuri, affermano i ricercatori. Spiegando ulteriormente la struttura RNAP di diversi batteri, gli scienziati possono progettare antibiotici che colpiscano ciascun patogeno in modo più selettivo ed efficace.