Uno studio ha attinto al genoma della pesca, creando reti di coespressione genica (GCN) su larga scala che prevedono le funzioni genetiche e semplificano il processo di selezione della pesca. Questo approccio innovativo affronta il complesso compito di identificare i geni legati ai tratti genetici desiderabili nelle pesche.
La genetica della pesca deve affrontare sfide quali lunghi tempi di intergenerazione e sistemi di trasformazione genetica limitati, che rendono complessa l’identificazione e la validazione dei geni. Gli approcci tradizionali spesso non riescono a prevedere le funzioni genetiche a causa di queste limitazioni.
Le reti di coespressione genica (GCN), che mappano le relazioni tra le espressioni genetiche, offrono una soluzione promettente. Sfruttando il principio della “colpevole per associazione”, i GCN possono prevedere le funzioni genetiche sulla base di modelli di espressione condivisi. A causa di queste sfide, esiste un urgente bisogno di strumenti innovativi come i GCN per migliorare la nostra comprensione della genetica delle pesche.
I ricercatori dell'Institut de Recerca i Tecnologia Agroalimentàries (IRTA) e del Centre de Recerca en Agrigenòmica (CRAG) hanno pubblicato uno studio sulla rivista Horticulture Research il 2 gennaio 2024. Lo studio introduce un nuovo strumento che sfrutta i GCN per prevedere le funzioni genetiche nelle pesche, rivoluzionando potenzialmente la selezione dei frutti.
Lo studio ha creato quattro GCN da 604 librerie Illumina RNA-Seq e ne ha valutato le prestazioni utilizzando un algoritmo di apprendimento automatico basato sul principio "colpevole per associazione". Tra le reti, COO300 ha mostrato le prestazioni migliori, comprendendo 21.956 geni. Per convalidare COO300, i ricercatori hanno condotto due casi di studio.
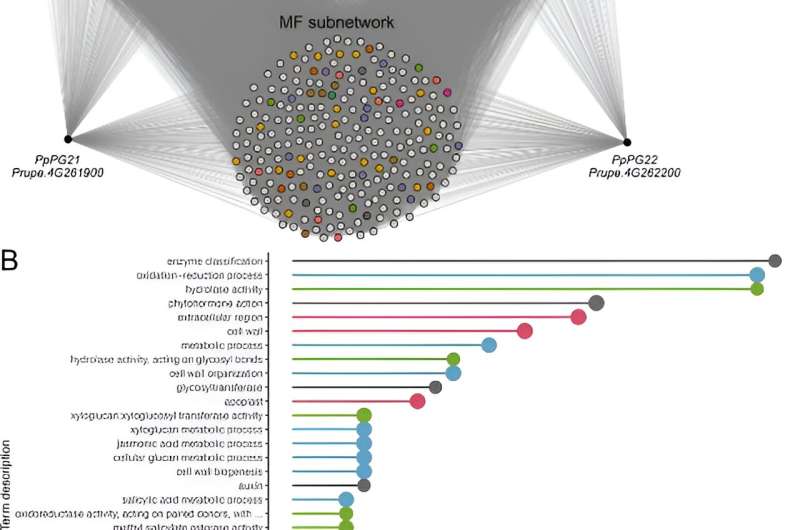
Nel primo caso studio sono stati analizzati i geni PpPG21 e PpPG22, coinvolti nel rammollimento del frutto della pesca. I geni co-espressi hanno formato la rete Melting Meat (MF), che è stata arricchita con termini relativi all'organizzazione della parete cellulare e alla segnalazione dei fitormoni. La rete MF includeva geni associati al disassemblaggio della parete cellulare e fitormoni legati alla maturazione, dimostrando la capacità di COO300 di prevedere accuratamente le funzioni genetiche.
Nel secondo caso di studio è stato esaminato il fattore di trascrizione PpMYB10.1, che regola l'accumulo di antociani. La rete del colore del frutto (FC) ha identificato i geni coinvolti nel metabolismo e nella regolazione degli antociani. Confrontando la rete FC con gli ortologi della vite, i ricercatori hanno identificato reti regolatorie conservate, convalidando ulteriormente le capacità predittive di COO300.
Il dottor Iban Eduardo, uno dei principali ricercatori del CRAG, ha dichiarato:"Lo sviluppo della rete COO300 rappresenta un passo avanti significativo nella genetica delle pesche. Predicendo accuratamente le funzioni genetiche, questo strumento non solo migliora la nostra comprensione della biologia delle pesche, ma apre anche la strada a strategie di allevamento più mirate ed efficienti."
Lo studio offre ai coltivatori un potente strumento per valorizzare le varietà di pesche. Decodificando le funzioni genetiche, si prevede che PeachGCN v1.0 e i suoi script, accessibili a tutti, favoriranno progressi nel sapore, nella longevità e nella nutrizione delle pesche. Questa conoscenza genomica è foriera di cambiamento e segnala uno spostamento verso la selezione basata sui dati in agricoltura.
Ulteriori informazioni: Felipe Pérez de los Cobos et al, Esplorazione di reti di coespressione genica su larga scala nella pesca (Prunus persica L.):un nuovo strumento per prevedere la funzione genetica, Ricerca sull'orticoltura (2024). DOI:10.1093/ora/uhad294
Informazioni sul giornale: Ricerca sull'orticoltura
Fornito dall'Università di Agraria di NanJing