Quando due animali hanno lo stesso aspetto, mangiano allo stesso modo, si comportano allo stesso modo e vivono in ambienti simili, ci si potrebbe aspettare che appartengano alla stessa specie.
Tuttavia, un minuscolo zooplancton che sfiora la superficie dell’oceano con microscopiche particelle di cibo mette in discussione questa ipotesi. Ricercatori dell’Università di Osaka, dell’Università di Barcellona e dell’Okinawa Institute of Science and Technology (OIST) hanno analizzato il genoma di Oikopleura dioica del Mare interno di Seto, del Mediterraneo e dell’Oceano Pacifico attorno alle isole di Okinawa, e così facendo, hanno hanno sollevato numerose domande sulla speciazione e sul ruolo della localizzazione dei geni nel genoma.
I loro risultati sono stati pubblicati su Genome Research . "Oikopleura sta aprendo nuove strade nella ricerca genomica", afferma il dottor Charles Plessy dell'Unità di genomica e sistemi di regolamentazione dell'OIST e co-primo autore dell'articolo.
"Come animale modello, ci consente di studiare i meccanismi dei cambiamenti genomici in laboratorio mentre avvengono su larga scala e a grande velocità, il che rappresenta un'enorme opportunità."
L'Oikopleura dioica è un minuscolo zooplancton che abita la superficie dell'oceano in tutto il mondo e che viene utilizzato come organismo modello nella biologia dello sviluppo.
In quanto cordato, l'organismo condivide tratti genetici e di sviluppo chiave con i vertebrati, inclusa la presenza di una notocorda, che è un fascio nervoso centrale simile ad una corda come una colonna vertebrale, ma senza ossa. Inoltre, il suo genoma compatto, il più piccolo genoma animale non parassitario finora riportato, facilita l'analisi genomica su larga scala.
I ricercatori hanno lavorato su tre lignaggi di Oikopleura dioica campionati da tre mari di tutto il mondo, ma sebbene le caratteristiche morfologiche, comportamentali ed ecologiche dei lignaggi siano praticamente le stesse, i genomi differiscono enormemente.
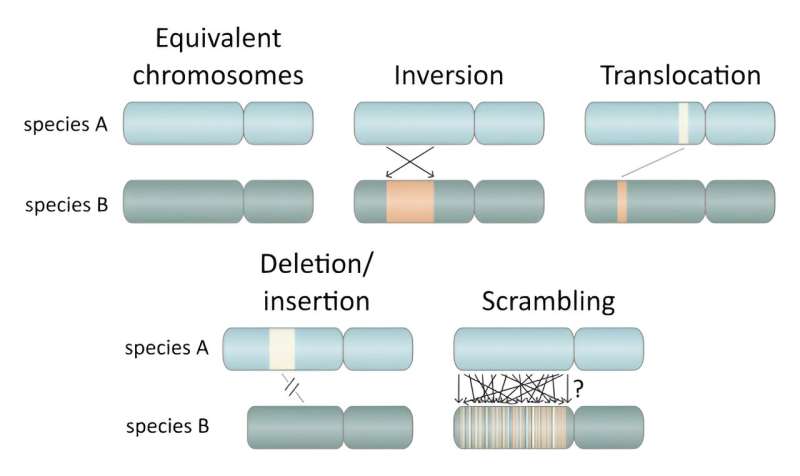
Pensate al genoma come ad un linguaggio condiviso tra tutti i membri di una singola specie, immagazzinato nel nucleo di ogni cellula e contenente l’insieme completo di materiale genetico necessario a formare quella specie. Come la grammatica determina la disposizione delle parole per trasmettere significati specifici, così anche le unità di base dell'informazione nel genoma, i geni, sono regolate l'una in relazione all'altra quando vengono trascritte e tradotte negli elementi costitutivi fondamentali della vita, le proteine. .
La regolazione genetica coinvolge molteplici fattori che influiscono sull'attivazione o sulla velocità della trascrizione genetica, come altri geni, molecole nella cellula, ormoni e molti altri.
Ciò che lascia perplessi nel genoma dell'Oikopleura dioica è che le lingue dei tre lignaggi non sembrano corrispondere, nonostante abbiano caratteristiche fisiche quasi identiche. Cioè, il "significato" prodotto dai loro geni è per lo più lo stesso, mentre i linguaggi genomici sono molto diversi tra loro.
I ricercatori usano il termine "scrambling" per descrivere il fenomeno osservato nell'Oikopleura dioica, un termine che ha origine nella linguistica per denotare un fenomeno in cui le frasi vengono formulate utilizzando una varietà di ordini di parole diversi senza alcun cambiamento di significato.
Anche se questo fenomeno non si verifica in inglese (ma si verifica in giapponese e in altre lingue), un esempio inglese sarebbe se la frase "il genoma di Oikopleura dioica è altamente codificato" potesse essere riorganizzata in "Oikopleura dioica altamente codificato il genoma di è" senza cambiamento di significato. Sebbene i riarrangiamenti genomici siano comuni a tutte le specie e la codifica del genoma sia stata osservata in alcune specie per un periodo di tempo molto lungo, Oikopleura dioica supera ciò che in precedenza si riteneva possibile.
Evoluzione a una velocità vertiginosa
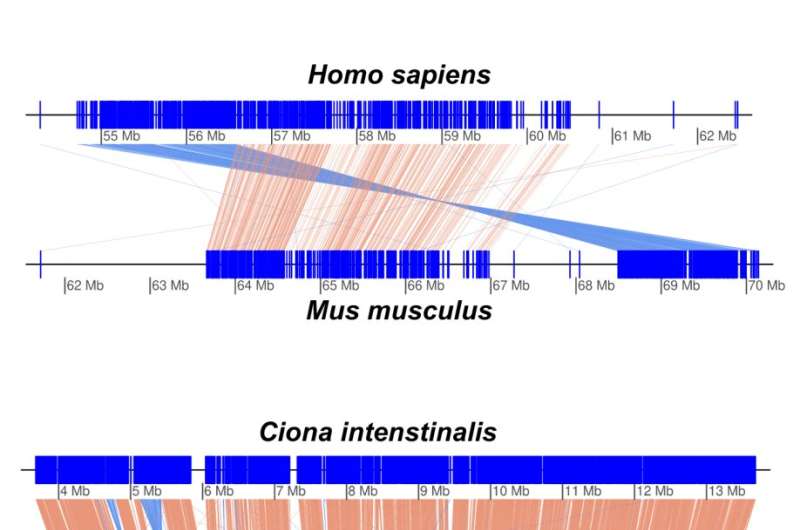
I ricercatori hanno confrontato le sequenze genetiche dei tre lignaggi, il che li ha portati a stimare che condividessero un antenato comune circa 25 milioni di anni fa, con i lignaggi di Barcellona e Osaka che erano più strettamente correlati rispetto al lignaggio di Okinawa, essendosi divergenti circa 7 milioni di anni. fa. Per fare un confronto, gli esseri umani si sono differenziati dai topi 75-90 milioni di anni fa.
Dalle loro analisi filogenetiche, i ricercatori hanno stimato il tasso di riarrangiamenti genomici per le diverse specie come misura quantificabile della velocità con cui si evolvono. Da ciò, i ricercatori hanno scoperto che il tasso di Oikopleura dioica è più di dieci volte superiore a quello di specie comparabili di ascidie di Ciona.
Come afferma il dottor Michael J. Mansfield dell'unità e co-primo autore dell'articolo:"L'Oikopleura è uno degli animali che si evolvono più rapidamente al mondo. Gli animali, in particolare i cordati, normalmente non riorganizzano i loro genomi a questo punto". , a questa velocità."
Con tutta questa confusione genomica che avviene tra i lignaggi dell'Oikopleura dioica, da una prospettiva genomica è sconcertante che possano mantenere caratteristiche così simili.
"I nostri risultati suggeriscono che, sebbene l'organizzazione genomica sia importante, soprattutto per qualcosa di così complesso come gli esseri umani, non dovremmo dimenticare i singoli geni", suggerisce il dottor Plessy. Lo studio dei geni e dei genomi può offrire due diverse prospettive sullo stesso fenomeno:come spiega il dottor Mansfield:"Ci sono scienziati che studiano l'anatomia e altri che studiano i singoli neuroni, ma entrambi rispondono a domande sul cervello".
La codifica del genoma pone importanti domande sull’evoluzione e sulla classificazione della vita in specie. Da un lato, i ricercatori mostrano che, anche se i tre lignaggi di Oikopleura dioica sono praticamente identici morfologicamente e funzionalmente, i loro genomi sono estremamente confusi, il che potrebbe suggerire che appartengano a specie diverse, anche se i ricercatori sottolineano che la loro intenzione non è quella di classificarli qui. D'altra parte, l'espressione genica confusa ma analoga potrebbe mettere in guardia contro un eccessivo affidamento alla genomica per la classificazione delle specie.
Alla fine, però, "le specie non hanno bisogno di noi. Se elimini gli esseri umani, gli animali restano gli stessi:non importa come li classifichiamo", come dice il dottor Plessy. Invece il concetto di specie è fluido, a seconda che sia per scopi di conservazione, per legislazione, come microbiologo o zoologo, o qualunque sia il motivo. "La domanda 'che cos'è una specie?' si può rispondere con un'altra domanda:perché lo chiedi?"
Per il dottor Plessy, il dottor Mansfield e i loro collaboratori in tutto il mondo, questo articolo è il culmine di un lungo processo di coltivazione di diversi lignaggi di Oikopleura dioica e di sviluppo di strumenti bioinformatici in grado di analizzare i loro genomi caotici. Il professor Nicholas Luscombe, capo dell'unità dell'OIST, è ottimista riguardo al potenziale di ricerca dello studio e degli animali.
"Inizialmente pensavamo che tutti gli Oikopleura avessero genomi simili, ma siamo rimasti stupiti nel vedere differenze così enormi con così tanta confusione tra di loro. Vogliamo utilizzare Oikopleura per saperne di più sulla natura dei riarrangiamenti genomici."
Questo è solo l’inizio:i ricercatori sono ben lungi dall’aver finito di studiare l’enigmatico zooplancton. "Abbiamo già imparato molto dall'Oikopleura, ma dobbiamo ancora esplorare l'intera portata della diversità delle specie su scala globale", afferma il dott. Plessy.
Il dottor Mansfield cita il grande biologo Jacques Monod con la frase "ciò che è vero per l'Escherichia coli è vero anche per l'elefante":con gli strumenti sviluppati per questo studio, i team possono ora rivolgere la loro attenzione ad altre specie. "Siamo arrivati a questo punto pensando che tutte le Oikopleura dioica fossero uguali, ma abbiamo dimostrato il contrario. Quanto spesso è vero per altre specie, e quanto c'è ancora da sapere sui meccanismi di rimescolamento del genoma?"
Ulteriori informazioni: Charles Plessy et al, Ricomposizione genomica estrema nelle specie planctoniche marine Oikopleura dioicacriptiche, Ricerca sul genoma (2024). DOI:10.1101/gr.278295.123
Informazioni sul giornale: Ricerca sul genoma
Fornito dall'Okinawa Institute of Science and Technology