Il potenziale antimicrobico dei sistemi CRISPR-Cas è promettente, ma il modo migliore per progettare o implementare le nucleasi CRISPR rimane poco compreso. Un team internazionale guidato dall'Istituto Helmholtz per la ricerca sulle infezioni basate su RNA (HIRI) di Würzburg ha ora colmato questa lacuna di conoscenze.
I ricercatori hanno condotto la prima interrogazione sistematica sugli antimicrobici CRISPR utilizzando batteri multiresistenti e ipervirulenti come casi di studio, rivelando ampie variazioni di efficacia che potrebbero essere previste tramite screening ad alto rendimento e apprendimento automatico. I loro risultati sono pubblicati sulla rivista Nucleic Acids Research .
La scoperta di composti antimicrobici come gli antibiotici convenzionali ha trasformato la medicina, consentendo il trattamento di infezioni un tempo ritenute incurabili. Tuttavia, il processo di sviluppo di nuovi agenti ha subito un rallentamento, mentre l’uso improprio degli antibiotici esistenti ha alimentato l’emergere della resistenza agli antibiotici. Di conseguenza, vi è una crescente necessità di nuovi mezzi per sradicare gli agenti patogeni.
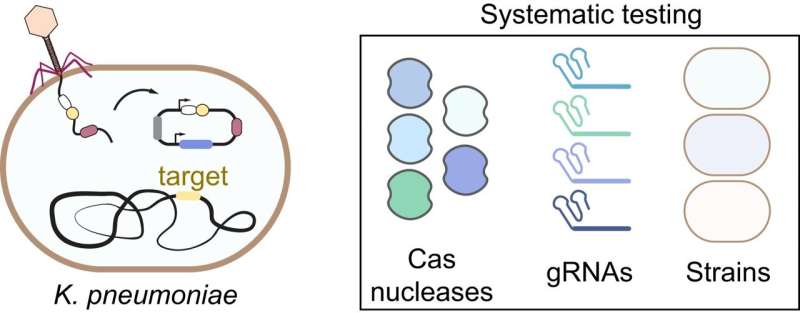
I sistemi CRISPR-Cas, meccanismi immunitari adattivi che i batteri impiegano per difendersi dall’invasione virale, offrono una soluzione distinta attraverso la loro capacità di eliminare selettivamente i microbi basandosi esclusivamente su sequenze genetiche. Tuttavia, ad oggi mancano studi sistematici per valutare l'efficacia di questi antimicrobici CRISPR, in particolare su diverse nucleasi, siti bersaglio e ceppi batterici.
Per affrontare questa lacuna, un team internazionale guidato dall’Istituto Helmholtz per la ricerca sulle infezioni basate sull’RNA (HIRI), una sede del Centro Helmholtz di Braunschweig per la ricerca sulle infezioni (HZI) in collaborazione con la Julius-Maximilians-Universität Würzburg (JMU), ha ora hanno intrapreso il primo interrogatorio completo di questi nuovi agenti. La loro ricerca si concentra sulla Klebsiella pneumoniae, un batterio noto per la sua associazione con la resistenza agli antibiotici.
"La Klebsiella pneumoniae offre un caso di studio particolarmente interessante dato che comprende numerosi ceppi con diverse caratteristiche di virulenza e resistenza", afferma Chase Beisel, capo del dipartimento di biologia sintetica dell'RNA presso l'HIRI e professore presso la Facoltà di medicina della JMU. Ha guidato lo studio internazionale in collaborazione con ricercatori dell'Institut Pasteur di Parigi, in Francia, dell'Università di Tel Aviv in Israele, dell'HZI e dell'Università di Toronto in Canada.
Il team ha unito le competenze nelle tecnologie CRISPR, nei batteri Klebsiella, nel rilascio di batteriofagi, negli schermi ad alto rendimento e nell'apprendimento automatico necessari per condurre uno studio di questa portata.
I sistemi CRISPR-Cas utilizzano un sofisticato meccanismo di difesa:un acido ribonucleico (RNA) CRISPR aiuta a rilevare regioni di un genoma estraneo, come DNA o RNA, per la scissione mirata. Successivamente, la nucleasi associata a CRISPR (Cas) taglia il suo bersaglio in modo simile a un paio di forbici molecolari.
Gli scienziati hanno scoperto che diverse nucleasi CRISPR mostrano efficacità molto diverse. Nei loro esperimenti, le nucleasi che prendevano di mira il DNA hanno mostrato prestazioni superiori rispetto a quelle che si concentravano sull'RNA.
Inoltre, diversi tipi di K. pneumoniae hanno mostrato variazioni nella loro sensibilità a un antimicrobico CRISPR, nonostante l’impiego di nucleasi identiche per colpire siti identici. Elena Vialetto, la prima autrice dello studio ed ex Ph.D. studente del laboratorio Beisel, afferma:"L'attività antimicrobica variabile tra batteri correlati è stata sorprendente dato l'uso degli stessi costrutti CRISPR. Abbiamo attribuito questa differenza al ripiegamento degli RNA CRISPR che guidano il targeting del DNA."
Beisel aggiunge:"Questo studio è il primo a dimostrare che l'efficacia antibatterica può variare anche tra ceppi correlati."
Per esplorare le caratteristiche che potrebbero migliorare il targeting tra vari ceppi, i ricercatori hanno condotto uno screening dell'intero genoma in diversi tipi di K. pneumoniae. Questo sforzo ha prodotto principi e parametri di progettazione per possibili antimicrobici CRISPR e ha facilitato l'addestramento di un algoritmo per prevederne l'efficacia.
Il team si è anche avventurato nella fase successiva dello sviluppo dell'agente attivo, ovvero la consegna. I ricercatori hanno utilizzato i batteriofagi come veicoli per gli antimicrobici CRISPR, che hanno dotato di fibre di coda modificate per aumentare la portata del carico CRISPR.
Questo studio getta le basi per l'ulteriore sviluppo di CRISPR come mezzo per prevenire o curare le infezioni resistenti agli antibiotici.
"Ci auguriamo che questo lavoro dia maggiore visibilità all'uso di CRISPR come antimicrobico ad spettro personalizzato nella lotta in corso contro la resistenza agli antibiotici", conclude Beisel.
Ulteriori informazioni: Elena Vialetto et al, L'interrogazione sistematica degli antimicrobici CRISPR in Klebsiella pneumoniae rivela caratteristiche nucleasi, guida e dipendenti dal ceppo che influenzano l'attività antimicrobica, Ricerca sugli acidi nucleici (2024). DOI:10.1093/nar/gkae281
Informazioni sul giornale: Ricerca sugli acidi nucleici
Fornito dal Centro Helmholtz per la ricerca sulle infezioni