Un gruppo di ricerca ha completato un assemblaggio su scala cromosomica di alta qualità del genoma del cartamo Chuanhonghua 1. Questo lavoro fa luce sulle basi genetiche di tratti cruciali come la produzione di acido linoleico (LA) e giallo idrossizafferano A (HSYA). Stabilisce un nuovo precedente per il miglioramento delle colture e gli studi di genomica funzionale.
Il cartamo (Carthamus tinctorius L.), una pianta nota per i suoi fiori vivaci e i semi ricchi di sostanze nutritive, è coltivata da migliaia di anni nella fertile regione della mezzaluna. È molto apprezzato per il suo olio, ricco di LA; e i suoi fiori, che contengono HSYA, sostanze con ampie applicazioni industriali e medicinali. Nonostante il suo valore, la ricerca genetica sul cartamo è stata limitata dalla conoscenza limitata del suo genoma.
Uno studio apparso su Horticulture Research chiarisce le basi genetiche del cartamo, aprendo così la strada a strategie di selezione mirate.
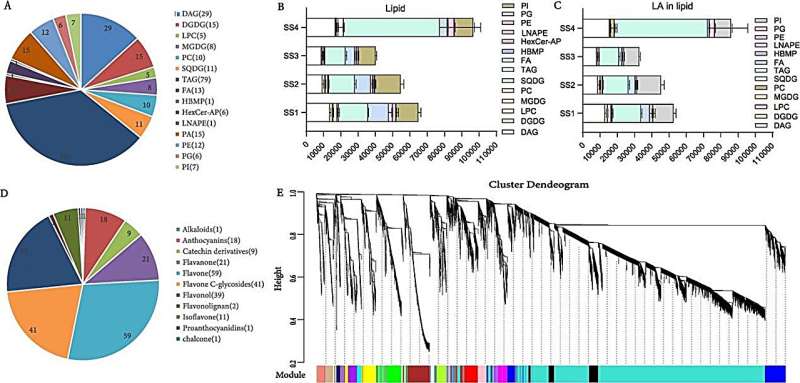
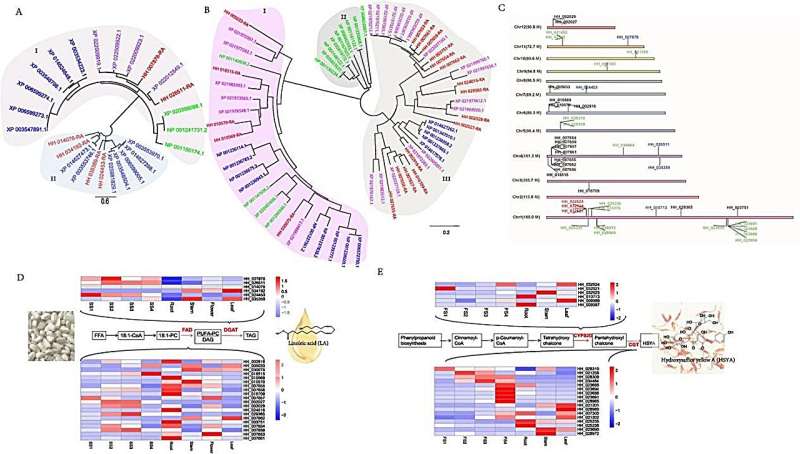
Il team di ricerca ha utilizzato una strategia di sequenziamento integrata che combina le tecnologie Illumina, Oxford Nanopore e Hi-C per ottenere un assemblaggio del genoma di alta qualità di 1,17 Gb con contigui assegnati a 12 cromosomi, riflettendo una copertura di circa 100 volte rispetto alla dimensione stimata del genoma. Questo assemblaggio ha facilitato l'identificazione di un recente evento di duplicazione dell'intero genoma, alla base degli estesi riarrangiamenti genomici e della storia evolutiva del cartamo, corroborati dall'analisi filogenetica utilizzando 274 geni a copia singola in 12 specie e da un esame dettagliato dell'espansione e della contrazione della famiglia genetica.
La profilazione metabolomica e trascrittomica nei diversi stadi di sviluppo di semi e fiori ha rivelato un lipidoma completo con una presenza predominante di triacilgliceroli (TAG) e uno spettro diversificato di metaboliti flavonoidi, rispettivamente, evidenziando le vie biosintetiche di composti chiave come LA e HSYA. Attraverso l'analisi genomica, i ricercatori hanno previsto 39.809 geni codificanti proteine, con sostanziali annotazioni rispetto ai database pubblici, e hanno identificato famiglie di geni specifici:diacilglicerolo aciltransferasi (DGAT) e desaturasi degli acidi grassi (FAD) per la biosintesi di LA e CYP e CGT per la biosintesi di HSYA. indicando il loro ruolo chiave in queste vie metaboliche.
In particolare, il risequenziamento di 220 linee di cartamo ha prodotto 7.402.693 SNP di alta qualità, facilitando uno studio di associazione su tutto il genoma (GWAS) che ha individuato SNP significativi associati ai tratti agronomici, in particolare al contenuto di olio e al colore dei fiori. Questa analisi GWAS, insieme alla verifica funzionale del gene candidato HH_034464 (CtCGT1), implicato nella biosintesi di HSYA, ha sottolineato il potenziale dei marcatori molecolari nel migliorare i programmi di allevamento per i tratti desiderati. I test funzionali hanno confermato il ruolo di CtCGT1 nella glicosilazione dei flavonoidi, cruciali per la produzione di HSYA, facendo così luce sulle basi genetiche dei principali tratti metabolici del cartamo.
Il dottor John Smith, un esperto di genomica vegetale, ha elogiato il risultato, affermando:"Questo completo assemblaggio del genoma è una pietra miliare nella ricerca sul cartamo. Non solo migliora la nostra comprensione dell'evoluzione delle piante, ma fornisce anche una ricca risorsa per identificare i geni responsabili delle principali tratti agricoli e medicinali."
Fornendo una base per la selezione molecolare del cartamo, questa ricerca offre il potenziale per migliorare la resilienza delle colture, il contenuto nutrizionale e l’efficacia terapeutica. Guardando al futuro, la sfida sta nello sfruttare questa vasta risorsa genomica per sviluppare varietà di cartamo superiori che soddisfino le esigenze di un clima globale in cambiamento e di una popolazione in crescita.
L'assemblaggio del genoma su scala cromosomica del cartamo Chuanhonghua 1 rappresenta un passo avanti fondamentale nell'esplorazione genetica di questa preziosa coltura. Questa ricerca non solo apre nuove strade per l'estrazione e la selezione dei geni funzionali del cartamo, ma ha anche implicazioni più ampie per l'industria agricola e farmaceutica.
Ulteriori informazioni: Jiang Chen et al, Gli studi sull'intero genoma e sull'associazione dell'intero genoma migliorano i principali tratti agricoli del cartamo per uso industriale e medicinale, Ricerca sull'orticoltura (2023). DOI:10.1093/ora/uhad197
Informazioni sul giornale: Ricerca sull'orticoltura
Fornito dall'Università di Agraria di NanJing