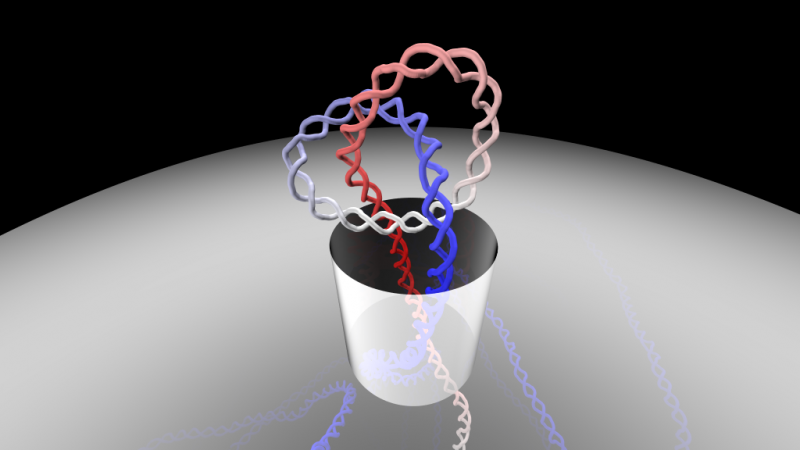
Come possono lunghi filamenti di DNA, che hanno una struttura contorta e molto annodata, riescono a passare attraverso i minuscoli pori di vari sistemi biologici? Questa è l'affascinante domanda rivolta da Antonio Suma e Cristian Micheletti, ricercatori della Scuola Internazionale Superiore di Studi Avanzati (SISSA) di Trieste che hanno utilizzato simulazioni al computer per studiare le opzioni a disposizione del materiale genetico in tali situazioni. Lo studio è stato appena pubblicato su PNAS , la rivista della National Academy of Sciences degli Stati Uniti. Credito:Antonio Suma, SISSA
Chiunque sia stato su una barca a vela sa che fare un nodo è il modo migliore per fissare una corda a un gancio ed evitare che scivoli. Allo stesso modo, i nodi nei fili per cucire impediscono che scivolino attraverso due pezzi di tessuto. Come, poi, possono lunghi filamenti di DNA, che hanno una struttura contorta e molto annodata, riescono a passare attraverso i minuscoli pori di vari sistemi biologici? Questa è l'affascinante domanda rivolta da Antonio Suma e Cristian Micheletti, ricercatori della Scuola Internazionale Superiore di Studi Avanzati (SISSA) di Trieste, che ha utilizzato simulazioni al computer per studiare la dinamica della molecola in tali situazioni. Lo studio è stato appena pubblicato su PNAS , la rivista della National Academy of Sciences degli Stati Uniti.
"Il nostro studio computazionale fa luce sulle ultime scoperte sperimentali sulla manipolazione del DNA annodato, e aggiunge elementi interessanti e inaspettati, “Spiega Micheletti. “Abbiamo prima osservato come i filamenti di DNA annodati passano attraverso minuscoli pori con un diametro di circa 10 nanometri (10 miliardesimi di metro). Il comportamento osservato nelle nostre simulazioni era in buon accordo con le misurazioni sperimentali ottenute da un gruppo di ricerca internazionale guidato da Cees Dekker, che sono stati pubblicati solo pochi mesi fa in Biotecnologie naturali . Questi esperimenti avanzati e sofisticati hanno segnato un punto di svolta per la comprensione dell'annodatura del DNA. Però, gli esperimenti attuali non sono in grado di rilevare come i nodi del DNA passino effettivamente attraverso il poro stretto.
"Infatti, il fenomeno si verifica su una scala spaziale minuscola, inaccessibile ai microscopi. Questo è il motivo per cui il nostro gruppo ha fatto ricorso a quello che il grande biofisico tedesco Klaus Schulten ha chiamato "il microscopio computazionale, ' questo è, simulazioni al computer".
Suma e Micheletti spiegano:"Le simulazioni hanno rivelato che il passaggio del nodo può avvenire in due modi distinti:uno dove il nodo è stretto, e l'altro dove il nodo è più delocalizzato. In entrambi i casi, il nodo non solo passa per il poro, ma lo fa in pochissimo tempo".
Inoltre, il nodo di solito passa nelle fasi finali della traslocazione, quando la maggior parte del filamento di DNA è già passato. "Ma c'è qualcosa di più che è controintuitivo, " affermano gli autori. "La dimensione del nodo, che sia piccolo o grande, non sembra influenzare di molto il tempo di ostruzione dei pori. Quest'ultimo dipende invece dalla velocità di traslocazione, quale, a sua volta, dipende dalla posizione iniziale del nodo lungo il filamento." Questi risultati, dicono i ricercatori, dovrebbe aiutare la progettazione di futuri esperimenti che sondano l'annodamento spontaneo del DNA, un luogo ancora in gran parte inesplorato, soprattutto per quanto riguarda la dimensione dei nodi del DNA.
Migliorare la nostra attuale comprensione dei nodi nelle molecole biologiche è importante per chiarire le loro implicazioni in contesti biologici così come in quelli applicativi, come il sequenziamento del DNA mediante nanopori. Suma e Micheletti sperano che le promettenti direzioni suggerite dal loro studio possano portare a un profilo più dettagliato e accurato dell'entanglement nel DNA, RNA e proteine.