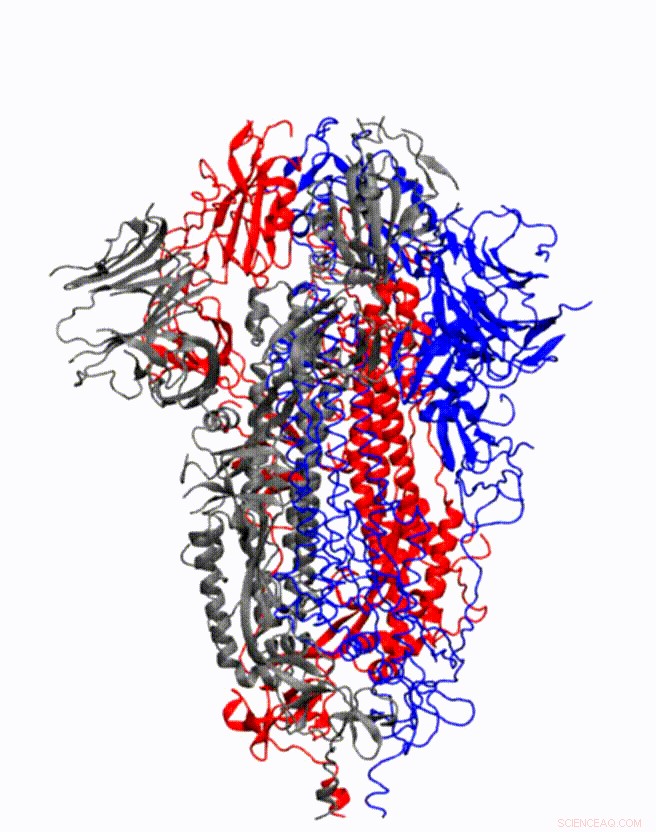
Animazione che mostra come la proteina spike del coronavirus cambia forma appena prima di legarsi al recettore delle cellule umane. Credito:illustrazione fornita da Mahmoud Moradi.
Il chimico computazionale Mahmoud Moradi svilupperà potenziato, Simulazioni 3D delle dinamiche molecolari delle glicoproteine del picco di coronavirus per ottenere una migliore comprensione di come il virus si lega alle cellule umane.
La mappatura di come queste proteine subiscono cambiamenti conformazionali per legarsi ai recettori delle cellule ospiti è fondamentale per lo sviluppo di vaccini e terapie contro il coronavirus. Le simulazioni sono particolarmente importanti perché un quadro per la progettazione di farmaci richiederà dinamiche, visualizzazioni tridimensionali delle strutture e del comportamento delle cellule, piuttosto che un'immagine statica.
"Come con altri virus, un passaggio cruciale nel processo di infezione da coronavirus è l'ingresso del virus, " disse Moradi, assistente professore al J. William Fulbright College of Arts and Sciences. "Con i coronavirus, sappiamo che queste glicoproteine spike mediano l'ingresso nella cellula umana. Entrambi SARS-CoV-2, la causa del COVID-19, e SARS-CoV, la causa dell'epidemia di SARS 2002-2003, hanno proteine spike che si attaccano allo stesso recettore nelle cellule umane".
Il lavoro di Moradi fa parte del COVID-19 High Performance Computing Consortium, una collaborazione di governo, partner industriali e accademici si sono concentrati sulle risorse informatiche per la ricerca COVID-19. Guidato dall'Ufficio per la politica scientifica e tecnologica della Casa Bianca, il Dipartimento dell'Energia degli Stati Uniti e IBM, i volontari del consorzio calcolano gratuitamente tempo e risorse su alcuni dei supercomputer più potenti del mondo.
Per eseguire le simulazioni, Moradi ha ottenuto l'accesso a Frontera, un supercomputer sponsorizzato dalla National Science Foundation ospitato presso l'Università del Texas ad Austin. Frontera è il più grande supercomputer di qualsiasi campus universitario.
Il progetto di Moradi beneficia di diversi recenti, modelli 3D ad alta risoluzione delle proteine spike del coronavirus. Questi modelli possono essere utilizzati come strutture iniziali per avviare simulazioni che consentiranno l'analisi dei meccanismi dettagliati delle proteine e del loro comportamento all'ingresso del virus. Migliorato, simulazioni dettagliate di tali dinamiche molecolari forniranno un quadro completo dei cambiamenti strutturali delle proteine, e come si legano all'enzima di conversione dell'angiotensina 2, il recettore specifico delle cellule umane.
La ricerca di Moradi si colloca all'incrocio tra biologia, fisica, chimica, matematica, statistica e informatica. Le sue simulazioni biomolecolari e teorie computazionali spiegano come le proteine, le molecole del cavallo di battaglia delle cellule, funzione a livello molecolare. Il suo lavoro migliora i modelli geometrici per descrivere come le proteine cambiano la loro forma e come questi cambiamenti influenzano il comportamento di una proteina. A febbraio, ha ricevuto un $ 650, 000 Premio per lo sviluppo della carriera iniziale della Facoltà della National Science Foundation per questo lavoro.