Un team di ricercatori ha recentemente dimostrato l'utilità di utilizzare un sistema di screening precedentemente stabilito per sondare le interazioni tra piccole molecole e RNA. Il loro studio è pubblicato in Communications Chemistry .
Il team era guidato dal professor Hirohide Saito (Dipartimento di frontiere delle scienze della vita, CiRA), dal dottor Kaoru R. Komatsu (un ex studente di dottorato al CiRA), dal professore associato Kazumitsu Onizuka e dal professor Fumi Nagatsugi (Istituto di ricerca multidisciplinare per i materiali avanzati, Università di Tohoku).
Dai recenti vaccini a mRNA SARS-CoV-2 per combattere la pandemia di COVID-19 a risdiplam, un modificatore di splicing dell'RNA approvato dalla Food and Drug Administration statunitense per l'atrofia muscolare spinale, la parola "RNA" è entrata nel linguaggio comune in quanto rappresenta sia nuove classi di agenti terapeutici e bersagli farmacologici.
Tuttavia, la nostra comprensione di come le varie sequenze di RNA e, a loro volta, le strutture determinano le interazioni con piccole molecole come composti farmaceutici o grandi biomolecole come le proteine rimane incompleta.
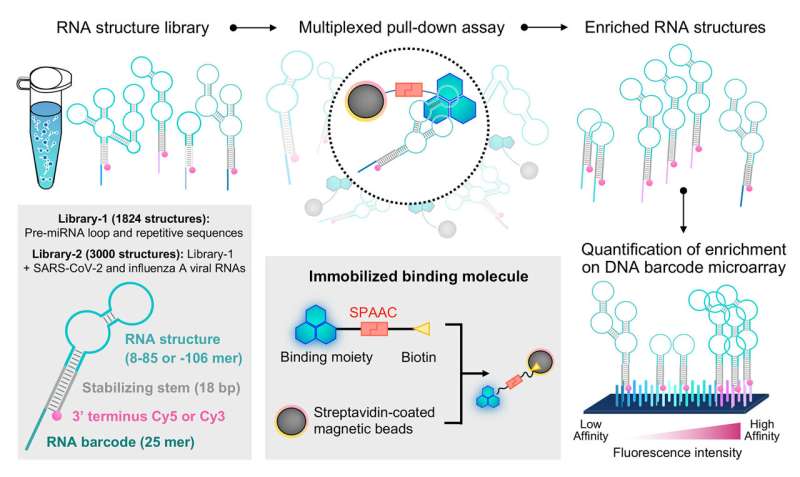
In uno studio precedente, il professor Saito e il suo gruppo di ricerca hanno progettato un sistema chiamato profilazione di elementi di RNA ripiegati con libreria di strutture, o FOREST, per esaminare i dettagli molecolari di come l'RNA interagisce con le proteine note che legano l'RNA.
Per questo nuovo studio, attraverso una collaborazione con ricercatori dell'Università di Tohoku, il team di ricerca congiunto ha illustrato come FOREST può essere utilizzato per analizzare piccole interazioni molecolari con l'RNA.
Il gruppo di ricerca ha innanzitutto convalidato l'applicabilità dell'approccio FOREST alle piccole molecole esaminando come una libreria di strutture di RNA interagisce con noti interattori di RNA molecolari piccoli:G-clamp e derivati dell'arancio tiazinico (TO).
I ricercatori hanno isolato le strutture di RNA legate al G-clamp da una libreria comprendente più di 1.800 sequenze di RNA derivate da pre-miRNA umani e altre sequenze ripetitive e di controllo. Poiché queste strutture di RNA sono collegate a un colorante fluorescente e a un codice a barre di RNA, potrebbero essere facilmente decodificate e quantificate da un microarray di DNA con sequenze complementari ai codici a barre di RNA per fornire un'analisi quantitativa di come le strutture di RNA interagiscono con la specifica piccola molecola di interesse.
Come previsto, hanno identificato il legame preferenziale con sequenze di RNA a filamento singolo e doppio contenenti guanosina (G) (rispettivamente ssRNA e dsRNA) mediante G-clamp. Dalla libreria della struttura dell'RNA, il team di ricerca ha scelto sequenze che mostrano un legame ad alta, intermedia o bassa affinità con il morsetto G per la convalida mediante un esperimento indipendente basato sulla fluorescenza che misura direttamente le costanti di dissociazione apparenti delle singole interazioni.
Sorprendentemente, hanno osservato una buona correlazione tra l'affinità di legame relativa stimata dall'approccio FOREST e le costanti di dissociazione apparenti determinate dal test di legame basato sulla fluorescenza, indicando l'elevata robustezza di questo metodo per quantificare le interazioni di piccole molecole-RNA.
Inoltre, mutando una specifica struttura del loop di RNA con più guanosine in posizioni diverse, hanno scoperto che il G-clamp non interagisce allo stesso modo con tutte le guanosine sul loop, ma che un contesto strutturale aggiuntivo può influenzare l'interazione.
Al contrario, i derivati TO sono sonde comunemente utilizzate per i test di spostamento dell'indicatore fluorescente (FID). I ricercatori hanno poi mescolato TO e TO-3 separatamente con una libreria di strutture di RNA espansa contenente sequenze aggiuntive derivate dagli RNA del virus SARS-CoV-2 e dell'influenza A per caratterizzare meglio i derivati TO per le misurazioni dell'RNA.
Come previsto, mentre non c'era alcuna correlazione tra i profili di legame delle strutture di RNA che interagiscono con il G-clamp e i derivati TO, TO e TO-3 condividevano profili di legame simili con alcune piccole distinzioni.
Ulteriori confronti tra TO-N3 , TO-N3 -2 e TO-3-N3 ha rivelato che la posizione del linker ha un'influenza modesta sui profili di legame dell'RNA. Inoltre, sulla base di questi profili di legame, i ricercatori hanno individuato alcune preferenze posizionali di base e di anello che i derivati TO hanno quando interagiscono con le strutture di RNA.
Il gruppo di ricerca ha inoltre esteso la propria analisi dei derivati del TO confrontando le affinità di legame relative determinate da FOREST con le costanti di dissociazione apparenti misurate per gli indicatori nucleici fluorescenti disponibili in commercio, TO-PRO-1 e TO-PRO-3, mediante il test di legame basato sulla fluorescenza .
Attraverso questa analisi, hanno rivelato che mentre TO-N3 -2 può rappresentare in modo più accurato il profilo vincolante di TO-PRO1 rispetto a TO-3-N3 , entrambi TO-N3 -2 e TO-3-N3 simula TO-PRO-3 quasi altrettanto bene, fornendo così approfondimenti strutturali cruciali per migliorare gli accoppiamenti di RNA target e indicatori fluorescenti per i test FID.
Utilizzando i profili di legame determinati per i derivati TO, il gruppo di ricerca ha selezionato combinazioni di indicatori fluorescenti (TO-PRO-1 o TO-PRO-3) e sequenze pre-miRNA che in precedenza avevano dimostrato di essere disregolate nei tumori con affinità di legame intermedie per i test FID.
Come prova di concetto, i ricercatori hanno esaminato una libreria chimica disponibile in commercio con 118 composti per identificare piccole molecole in grado di interagire con i pre-miRNA associati alla malattia. Attraverso questo sforzo, hanno identificato baicaleina (Bai), miricetina (Myr), cheleritrina cloruro (Che) e AS 602801 (AS) come composti candidati di successo. Mentre è noto che Myr e Che legano DNA e RNA, questa è stata la prima dimostrazione dell'AS come interattore dell'acido nucleico.
In particolare, i ricercatori hanno osservato risultati diversi quando TO-PRO-1 o TO-PRO-3 è stato utilizzato come indicatore fluorescente, suggerendo così che dovrebbero essere utilizzati indicatori distinti per evitare identificazioni false positive e negative. Un ulteriore esame dell'AS ha confermato il legame con diversi pre-miRNA umani di interesse, ma i ricercatori hanno anche notato che il composto mostra forti proprietà di illuminazione quando interagisce con l'RNA.
L'esame strutturale del composto suggerisce che contenga una frazione chimica probabilmente responsabile delle proprietà di illuminazione, rendendolo un composto di interesse per un ulteriore sviluppo in un nuovo interattore di RNA e sonda fluorescente.
In questo studio, lo sforzo di ricerca congiunto illustra ancora una volta l'applicabilità della metodologia FOREST, non solo per ispezionare le interazioni RNA-proteine ma anche per studiare i dettagli più fini delle interazioni tra RNA e piccole molecole.
Considerato l'ampio potenziale dell'RNA come nuovo approccio terapeutico nella medicina di prossima generazione, la capacità di caratterizzare sistematicamente le interazioni di piccole molecole-RNA su larga scala avrà un impatto enorme sulla ricerca di base sull'RNA e sulla traduzione di tale conoscenza in terapie.
Ulteriori informazioni: Ryosuke Nagasawa et al, Analisi su larga scala di interazioni di piccole molecole-RNA utilizzando librerie di strutture di RNA multiplex, Chimica delle comunicazioni (2024). DOI:10.1038/s42004-024-01181-8
Informazioni sul giornale: Chimica delle comunicazioni
Fornito dall'Università di Kyoto