
Credito:DeepMind
I giocatori applaudono, dì parole come Whoo, colpisci i coltelli di plastica sul tavolo e goditi i migliori fine settimana con l'intelligenza artificiale come atto principale, grazie all'intelligenza artificiale scatenata nei giochi.
WIRED UK editore di scienze, Matt Reynolds, ha esaminato l'impatto di DeepMind sulle pietre miliari dell'IA:"Ha superato i campioni di Go, ha battuto i giocatori professionisti di StarCraft e ha rivolto la sua attenzione agli scacchi e allo shogi."
Lasciamo che i giochi continuino, ma le cose serie devono brillare seriamente. In breve, possiamo ammirare che l'utilizzo dell'intelligenza artificiale a scopo di scoperta scientifica è diventato particolarmente vivo e vegeto grazie alla ricerca presso DeepMind.
Gli osservatori tecnologici hanno commentato questa settimana i documenti di ricerca che mostrano i punti di forza dell'intelligenza artificiale. "Man mano che l'intelligenza artificiale matura come campo (ed esaurisce i videogiochi da conquistare) probabilmente molti dei suoi risultati saranno simili a questi:solidi miglioramenti in importanti domini di ricerca".
Un documento di ricerca pubblicato su Natura ha fatto notizia immediata come un documento su come l'intelligenza artificiale può essere utilizzata per prevedere il ripiegamento delle proteine. VentureBeat riferito a sfide formidabili che si avvicinano sempre di più agli obiettivi.
Il documento è "Previsione migliorata della struttura proteica utilizzando i potenziali dell'apprendimento profondo, "L'articolo è stato pubblicato online il 15 dicembre in Natura .
Il sistema DeepMind si chiama AlphaFold. Reynolds ha avuto una nota interessante su AlphaFold e il set di dati:"Il campo del ripiegamento delle proteine è anche ben impostato per addestrare agenti artificialmente intelligenti. Ha un ampio set di dati:la banca dati proteica, un repository della struttura 3-D e del corredo genetico di 150, 000 proteine, che è stato utilizzato per addestrare il sistema di previsione della struttura proteica di DeepMind, chiamato AlphaFold."
Il documento ha esplorato i risultati ottenuti finora da DeepMind nell'uso dell'intelligenza artificiale per prevedere il ripiegamento delle proteine. Il grosso problema del ripiegamento delle proteine è che, nel ruolo di Kelsey Piper in Vox osservato, è una "questione cruciale per lo sviluppo di nuovi farmaci".
Tre del team DeepMind, Andrea Senior, John Jumper e Demis Hassabis, ha pubblicato un post sul blog del 15 gennaio che spiega le strutture proteiche, la sfida del ripiegamento delle proteine e ciò che volevano ottenere.
"Come dimostrato dal paradosso di Levinthal, ci vorrebbe più tempo dell'età dell'universo conosciuto per enumerare casualmente tutte le possibili configurazioni di una tipica proteina prima di raggiungere la vera struttura 3-D, eppure le proteine stesse si piegano spontaneamente, entro millisecondi. Prevedere come queste catene si piegheranno nell'intricata struttura 3D di una proteina è il cosiddetto "problema del ripiegamento delle proteine", una sfida su cui gli scienziati hanno lavorato per decenni".
Il team ha dimostrato di poter addestrare una rete neurale per fare previsioni accurate "delle distanze tra coppie di residui, che trasmettono più informazioni sulla struttura rispetto alle previsioni di contatto."
Hanno parlato di ottimizzazione tramite "un semplice algoritmo di discesa del gradiente per generare strutture senza complesse procedure di campionamento".
Il grosso problema del grassetto proteico è che, disse Piper, è una "questione cruciale per lo sviluppo di nuovi farmaci". Come mai? E, perché le "forme" sono così importanti? "La forma che assumono le proteine predice con quali altre sostanze interagiranno, quindi comprendere il ripiegamento delle proteine è fondamentale per la scoperta di farmaci e potrebbe anche essere utilizzato per lo sviluppo di nuovi processi di produzione, " disse Piper.
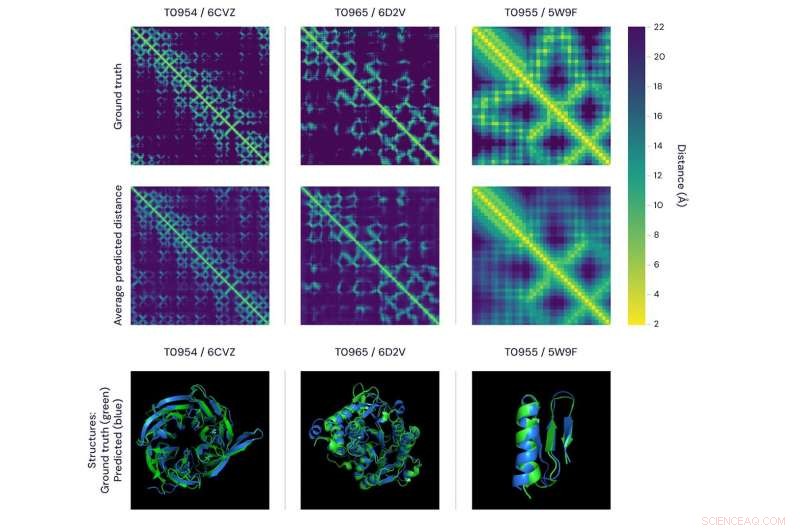
Due modi per visualizzare l'accuratezza delle previsioni di Alphafold. la figura in alto presenta le matrici di distanza per tre proteine. La luminosità di ciascun pixel rappresenta la distanza tra gli amminoacidi nella sequenza che comprende la proteina:più luminoso è il pixel, più la coppia è vicina. Nella riga in alto sono mostrati i veri, distanze determinate sperimentalmente e, nella riga in basso, la media delle distribuzioni di distanza previste da AlphaFold. È importante sottolineare che questi si abbinano bene sia su scala globale che locale. i pannelli inferiori rappresentano lo stesso confronto utilizzando modelli 3D, con le previsioni di AlphaFold (blu) rispetto ai dati veritieri (verde) per le stesse tre proteine. Credito:DeepMind
Il blog DeepMind ha affermato che "si ritiene che la forma di una proteina ne determini la funzione. Una volta compresa la forma di una proteina, il suo ruolo all'interno della cellula può essere indovinato, e gli scienziati possono sviluppare farmaci che funzionano con la forma unica della proteina".
AlphaFold potrebbe aiutare i ricercatori a comprendere meglio la funzione e il malfunzionamento delle proteine.
Una citazione degna di nota in VentureBeat dal capo del gruppo di bioinformatica dell'UCL David Jones, che ha consigliato il team di DeepMind su parti del progetto:
"Le tecniche sperimentali per determinare le strutture proteiche richiedono tempo e sono costose, quindi c'è un'enorme richiesta di algoritmi informatici migliori per calcolare le strutture delle proteine direttamente dalle sequenze geniche che le codificano, e il lavoro di DeepMind sull'applicazione dell'intelligenza artificiale a questo problema di vecchia data nella biologia molecolare è un netto progresso".
Quali tipi di tecniche sperimentali sono state lunghe e costose? Il blog ha menzionato la microscopia crioelettronica, risonanza magnetica nucleare e cristallografia a raggi X, ciascuno a seconda di tentativi ed errori, "che può richiedere anni di lavoro, e costa decine o centinaia di migliaia di dollari per struttura proteica".
Kyle Wiggers in VentureBeat :"Con AlphaFold, Il team di ricerca di DeepMind si è concentrato sul problema della modellazione di forme target da zero senza attingere a proteine risolte come modelli".
Come la Natura definisce il ripiegamento delle proteine:"Il ripiegamento delle proteine è il processo mediante il quale le proteine raggiungono la loro struttura terziaria funzionale matura (nativa), e spesso inizia in modo co-traduzionale. Il ripiegamento delle proteine richiede chaperon e spesso comporta la creazione graduale di strutture regolari secondarie e supersecondarie, vale a dire α-eliche e -fogli, che si piegano rapidamente, stabilizzato da legami idrogeno e ponti disolfuro, e poi la struttura terziaria."
Tornato a luglio, prima della pubblicazione del documento DeepMind, Natura aveva già preso atto che "la corsa per superare una delle più grandi sfide della biologia - predire le strutture 3-D delle proteine dalle loro sequenze di amminoacidi - si sta intensificando, grazie ai nuovi approcci di intelligenza artificiale (AI)".
Il post sul blog di DeepMind afferma di aver reso disponibile il codice (GitHub), e ha offerto il collegamento, per chiunque voglia saperne di più o replicare i propri risultati.
Nel loro post sul blog hanno affermato perché il loro sistema è importante:"Il nostro sistema, AlphaFold... si basa su decenni di ricerche precedenti utilizzando grandi set di dati genomici per prevedere la struttura delle proteine. I modelli 3D delle proteine generati da AlphaFold sono molto più accurati di quelli mai visti prima".
Andando avanti, il blog ha affermato che uno strumento come AlphaFold potrebbe aiutare i ricercatori sulle malattie rare a prevedere la forma di una proteina di interesse in modo rapido ed economico e potrebbe eventualmente aiutarli a contribuire alla scoperta efficiente di farmaci, riducendo i costi associati alla sperimentazione.
La ricerca sulle malattie rare non è l'unica area che può trarre vantaggio da ciò che hanno realizzato. Il blog parlava di inquinanti come plastica e petrolio; i progressi negli enzimi biodegradabili abilitati dalla progettazione delle proteine potrebbero aiutare a scomporre i rifiuti in modi più rispettosi del nostro ambiente.
"Infatti, i ricercatori hanno già iniziato a ingegnerizzare batteri per secernere proteine che renderanno biodegradabili i rifiuti, e più facile da elaborare, "secondo il blog.
© 2020 Scienza X Rete