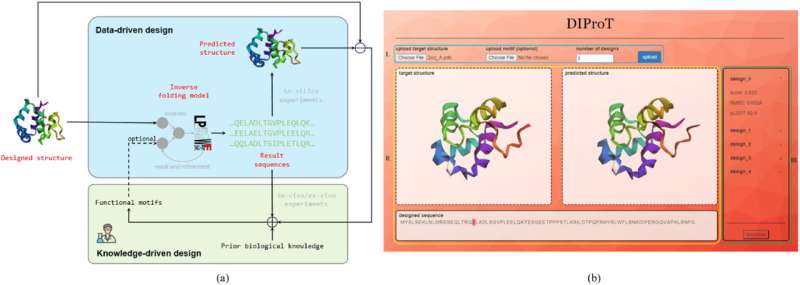
Gli scienziati hanno sviluppato DIProT, un kit di strumenti innovativo e facile da usare per la progettazione delle proteine. Il kit di strumenti utilizza un modello generativo profondo non autoregressivo per affrontare il problema del ripiegamento inverso delle proteine, integrando le competenze umane nel ciclo di progettazione per una progettazione proteica efficiente ed efficace.
La progettazione delle proteine, un aspetto cruciale delle scienze biologiche, prevede la creazione di sequenze di amminoacidi che si ripiegano nelle strutture proteiche desiderate. Questo processo, noto come problema del ripiegamento inverso delle proteine, è stato una sfida nel settore.
A tal fine, un team di ricercatori della Tsinghua University (THU) in Cina ha introdotto DIProT, un kit di strumenti interattivi per la progettazione di proteine che sfrutta un modello generativo profondo non autoregressivo per affrontare questo problema.
"Le proteine svolgono un ruolo cruciale in numerose funzioni biologiche", spiega l'autore corrispondente dello studio Xiaowo Wang, professore presso il Dipartimento di Automazione dell'Università di Tsinghua. "Sia la previsione della struttura di una determinata sequenza proteica, come esemplificato da AlphaFold, sia la progettazione di sequenze di amminoacidi conformi a una determinata struttura proteica pongono sfide uniche."
Per sviluppare DIProT, i ricercatori hanno integrato modelli di deep learning con competenze umane direttamente nel processo di progettazione, migliorando così l'efficienza e l'efficacia della progettazione delle proteine.
"L'approccio unico di DIProT consente agli utenti di specificare la struttura target e fissare le parti della sequenza che desiderano preservare, migliorando la flessibilità del processo di progettazione", aggiunge Wang. "Il toolkit incorpora anche un modello di previsione della struttura delle proteine per valutare i progetti in silico, formando un ciclo di progettazione virtuale che migliora significativamente l'efficienza della progettazione delle proteine."
Una delle caratteristiche principali di DIProT è la sua interfaccia utente grafica (GUI) intuitiva, che integra più algoritmi per facilitare un ciclo di progettazione di feedback rapido e intuitivo. La GUI consente agli utenti di interagire visivamente con i risultati della progettazione, aiutando la comprensione e l'interpretazione dei risultati.
Gli autori, che hanno pubblicato il loro studio sulla rivista Synthetic and Systems Biotechnology , ci aspettiamo che DIProT sia molto utile per attività pratiche di progettazione delle proteine. "Ci auguriamo che DIProT stimoli ulteriori ricerche sul campo e funga da strumento utile per affrontare sfide di progettazione proteica sempre più complesse e diversificate."
I ricercatori intendono perfezionare il loro modello di ripiegamento inverso e il loro kit di strumenti per affrontare in futuro sfide sempre più complesse e diversificate nella progettazione delle proteine.
Ulteriori informazioni: Jieling He et al, DIProT:un kit di strumenti interattivi basato sul deep learning per la progettazione efficiente ed efficace di proteine, biotecnologie sintetiche e di sistema (2024). DOI:10.1016/j.synbio.2024.01.011
Fornito da KeAi Communications Co., Ltd.