Appaiono all’improvviso e, come il coronavirus SARS-CoV-2, possono scatenare grandi epidemie:virus che nessuno aveva sul proprio radar. Non sono veramente nuovi, ma sono cambiati geneticamente. In particolare, lo scambio di materiale genetico tra diverse specie di virus può portare all'improvvisa comparsa di agenti patogeni minacciosi con caratteristiche notevolmente alterate.
Lo suggeriscono le attuali analisi genetiche effettuate da un team internazionale di ricercatori. I virologi del Centro tedesco per la ricerca sul cancro (DKFZ) erano responsabili dello studio su larga scala, pubblicato sulla rivista PLOS Pathogens .
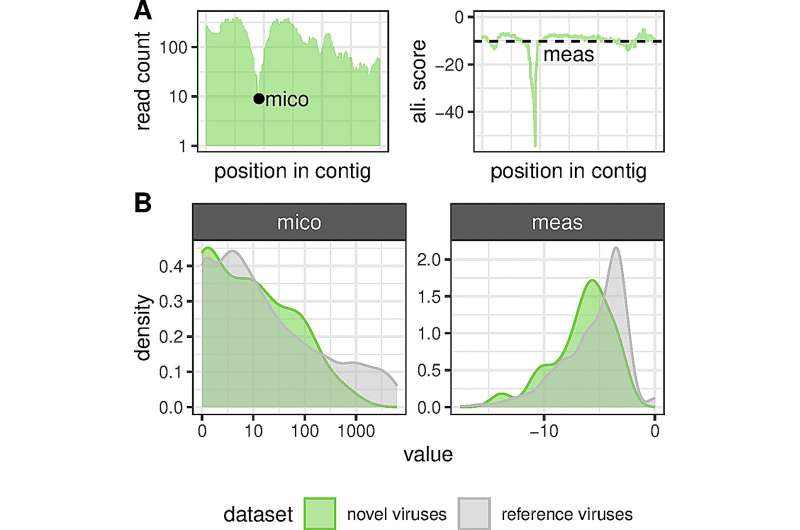
"Utilizzando un nuovo metodo di analisi computerizzata, abbiamo scoperto 40 nidovirus precedentemente sconosciuti in vari vertebrati, dai pesci ai roditori, inclusi 13 coronavirus", riferisce il leader del gruppo DKFZ Stefan Seitz. Con l'aiuto di computer ad alte prestazioni, il gruppo di ricerca, di cui fa parte anche il gruppo di lavoro di Chris Lauber del Centro Helmholtz per la ricerca sulle infezioni di Hannover, ha vagliato quasi 300.000 set di dati. Secondo il virologo Seitz, il fatto che ora possiamo analizzare quantità così grandi di dati contemporaneamente apre prospettive completamente nuove.
La ricerca sui virus è ancora nella sua relativa infanzia. Si conosce solo una frazione di tutti i virus presenti in natura, soprattutto quelli che causano malattie negli esseri umani, negli animali domestici e nelle colture. Il nuovo metodo promette quindi un salto di qualità nelle conoscenze per quanto riguarda il serbatoio naturale del virus. Stefan Seitz e i suoi colleghi hanno inviato dati genetici di vertebrati archiviati in database scientifici attraverso i loro computer ad alte prestazioni con nuove domande. Hanno cercato animali infetti dal virus per ottenere e studiare materiale genetico virale su larga scala. L'attenzione principale si è concentrata sui cosiddetti nidovirus, che includono la famiglia dei coronavirus.
I nidovirus, il cui materiale genetico è costituito da RNA (acido ribonucleico), sono diffusi nei vertebrati. Questo gruppo di virus ricco di specie ha alcune caratteristiche comuni che li distinguono da tutti gli altri virus a RNA e documentano la loro relazione. Per il resto, però, i nidovirus sono molto diversi tra loro, cioè in termini di dimensioni del loro genoma.
Una scoperta è particolarmente interessante per quanto riguarda la comparsa di nuovi virus:negli animali ospiti che vengono infettati contemporaneamente da virus diversi, durante la replicazione del virus può verificarsi una ricombinazione dei geni virali.
"A quanto pare, i nidovirus che abbiamo scoperto nei pesci scambiano spesso materiale genetico tra diverse specie di virus, anche oltre i confini familiari", afferma Seitz. E quando parenti lontani si "incrociano", ciò può portare alla comparsa di virus con proprietà completamente nuove. Secondo Seitz, tali salti evolutivi possono influenzare l'aggressività e la pericolosità dei virus, ma anche il loro attaccamento a determinati animali ospiti.
"Uno scambio genetico, come abbiamo riscontrato nei virus dei pesci, probabilmente avverrà anche nei virus dei mammiferi", spiega Seitz. I pipistrelli, che, come i toporagni, sono spesso infettati da un gran numero di virus diversi, sono considerati un vero e proprio crogiolo. Probabilmente il coronavirus SARS-CoV-2 si è sviluppato anche nei pipistrelli e da lì è passato all'uomo.
Dopo lo scambio genetico tra nidovirus, la proteina spike con cui i virus si agganciano alle cellule ospiti spesso cambia. Chris Lauber, primo autore dello studio, è riuscito a dimostrarlo mediante l'analisi degli alberi genealogici. La modifica di questa molecola di ancoraggio può modificare in modo significativo le proprietà dei virus a proprio vantaggio, aumentandone l'infettività o consentendo loro di cambiare ospite.
Un cambiamento di ospite, soprattutto dagli animali all’uomo, può facilitare notevolmente la diffusione del virus, come ha dimostrato con enfasi la pandemia del coronavirus. I “game-changer” virali possono apparire all’improvviso in qualsiasi momento, diventando una minaccia enorme e, se arriva il momento critico, innescando una pandemia. Il punto di partenza può essere un singolo animale ospite con doppia infezione.
Il nuovo processo informatico ad alte prestazioni potrebbe aiutare a prevenire la diffusione di nuovi virus. Permette una ricerca sistematica di varianti di virus potenzialmente pericolose per l'uomo, spiega Seitz.
Il ricercatore della DKFZ vede un'altra importante possibile applicazione per quanto riguarda il suo campo speciale di ricerca, la carcinogenesi associata ai virus:"Potrei immaginare che potremmo usare il nuovo High Performance Computing (HPC) per esaminare sistematicamente i virus dei malati di cancro o delle persone immunocompromesse. Noi sappiamo che il cancro può essere innescato da virus, l'esempio più noto è il papillomavirus umano. Ma probabilmente finora vediamo solo la punta dell'iceberg. Il metodo HPC offre l'opportunità di rintracciare i virus che, prima non rilevati, si annidano organismo umano e aumentano il rischio di tumori maligni."
Ulteriori informazioni: Chris Lauber et al, L'estrazione profonda del Sequence Read Archive rivela importanti innovazioni genetiche nei coronavirus e altri nidovirus dei vertebrati acquatici, PLOS Pathogens (2024). DOI:10.1371/journal.ppat.1012163
Informazioni sul giornale: Patogeni PLoS
Fornito dal Centro tedesco per la ricerca sul cancro