A causa dell'elevata trasparenza delle cellule, è molto difficile osservare gli organelli al loro interno. I biologi possono etichettare organelli specifici per l'osservazione attraverso la colorazione a fluorescenza. Ciò è in qualche modo analogo all'essere in un ambiente senza luce dove tutti sono vestiti interamente di nero, rendendo difficile trovare i propri amici. Facendo tenere in mano ai nostri amici un bastoncino fluorescente, possiamo localizzarli facilmente.
Una domanda interessante è:se l'angolo del bastoncino fluorescente tenuto dal mio amico rappresenta una sorta di segnale, come possiamo rilevare tali informazioni angolari?
Proprio come in questo puzzle, a causa della natura altamente trasparente delle cellule, è molto difficile osservare gli organelli in esse contenuti. Con la colorazione fluorescente, i biologi possono etichettare organelli specifici per l'osservazione. La maggior parte delle molecole fluorescenti appaiono come dipoli direzionali durante l'assorbimento o l'emissione.
L'orientamento dei fluorofori può rivelare importanti informazioni sulla struttura e sulla dinamica degli organelli associati. Anche la microscopia a polarizzazione a fluorescenza si è sviluppata come strumento indispensabile per studiare le caratteristiche di orientamento delle biomolecole.
Per superare la sfida della microscopia di polarizzazione a fluorescenza convenzionale limitata dalla diffrazione ottica, sono state proposte tecniche migliorate di microscopia di polarizzazione a fluorescenza a super risoluzione, come la microscopia di orientamento-localizzazione di singola molecola (SMOLM) e la modulazione di polarizzazione (ad esempio, SDOM, SPoD, ecc. ).
Tuttavia, dal punto di vista biotecnologico, nonostante il ruolo significativo dei filamenti biologici (ad esempio, filamenti di actina e microtubuli) nelle funzioni cellulari, mancano approcci con risoluzione dell'orientamento 3D e alta risoluzione spazio-temporale per studiarli in vivo.
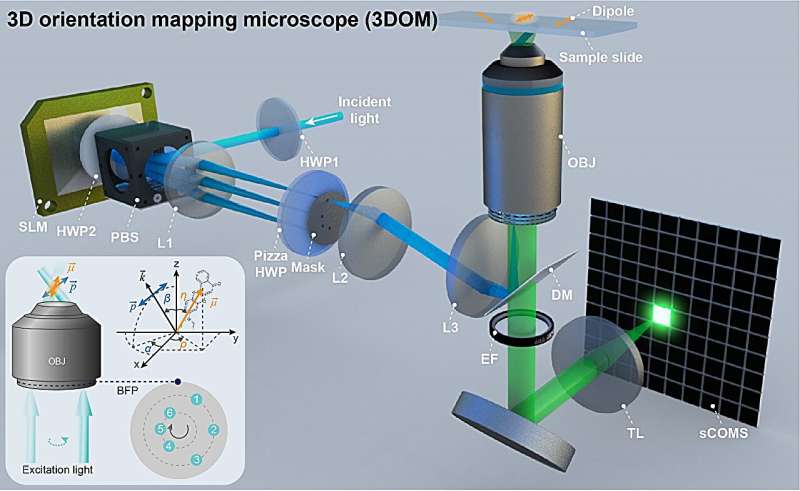
Per affrontare il problema della risoluzione dell'orientamento del dipolo, il gruppo di ricerca del professor Xi Peng dell'Università di Pechino ha sviluppato un metodo di mappatura dell'orientamento del dipolo 2D, SDOM, e una mappatura dell'orientamento del dipolo a super risoluzione con rilevamento ottico lock-in, OLID-SDOM. In PhotoniX , il gruppo di ricerca riporta un microscopio per la mappatura dell'orientamento 3D a super risoluzione chiamato 3DOM.
Il metodo 3DOM si basa sulla microscopia ad illuminazione strutturata polarizzata sviluppata dal gruppo di ricerca. Invertendo il principio dell'interferenza della doppia fenditura di Young e combinandolo con il principio dei percorsi luminosi reversibili, vengono utilizzati diversi angoli delle strisce per produrre fasci di primo ordine positivi e negativi in direzioni diverse.
Inoltre, è possibile produrre un'unica direzione di illuminazione inclinata semplicemente bloccando la corrispondente luce negativa del primo ordine. Proiettando questa inclinazione su diversi angoli dell'asse z e ricostruendo l'immagine utilizzando l'algoritmo FISTA, è possibile ottenere una risoluzione ad alta precisione dell'orientamento del dipolo combinando i coefficienti di modulazione della polarizzazione e i risultati della ricostruzione nello spazio reciproco.
Nel complesso, il metodo 3DOM proposto supera efficacemente i limiti della microscopia a fluorescenza polarizzata nella risoluzione spaziale e nella mappatura dell'orientamento 3D utilizzando l'imaging ad ampio campo.
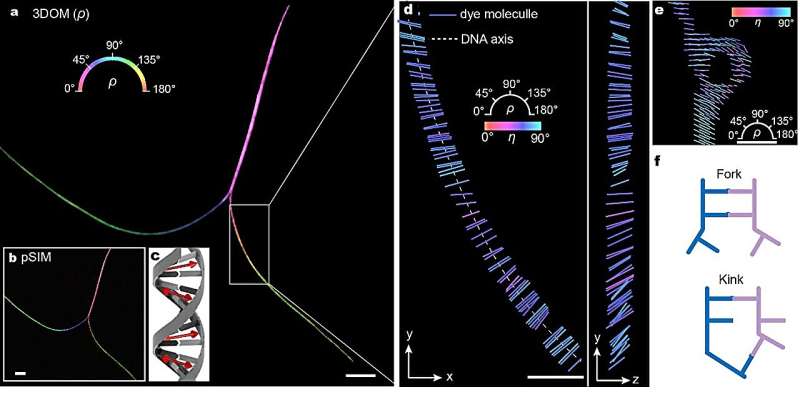
3DOM fornisce una comprensione più completa della struttura spaziale 3D delle molecole di fluoroforo. Ciò ci consente non solo di distinguere varie organizzazioni citoscheletriche (filamenti di actina e microtubuli), ma anche di ottenere preziose informazioni sulla compattezza del legame dei filamenti e sull'ordine delle strutture subcellulari.
Inoltre, 3DOM ha un potenziale significativo nella piegatura del DNA e nell'orientamento degli organelli membranosi. Uno dei principali vantaggi di 3DOM è la sua facilità di aggiornabilità ai sistemi widefield esistenti. La semplice implementazione, le informazioni accurate sull'orientamento del dipolo 3D e la risoluzione spaziotemporale superiore di 3DOM lo rendono adatto a un'ampia gamma di applicazioni, migliorandone l'accessibilità e l'usabilità in diversi contesti di ricerca.
Questo potente strumento consente ai ricercatori di svelare le intricate complessità della struttura subcellulare, della biomeccanica e della biodinamica, rivoluzionando la nostra comprensione dei processi cellulari. I ricercatori prevedono che 3DOM migliorerà la comprensione di una moltitudine di strutture biologiche e interazioni operative su scala nanometrica.
Ulteriori informazioni: Suyi Zhong et al, Mappatura tridimensionale dell'orientamento del dipolo con elevata risoluzione spazio-temporale utilizzando la modulazione di polarizzazione, PhotoniX (2024). DOI:10.1186/s43074-024-00127-6
Fornito dall'Università di Pechino