Un gruppo di ricerca ha sostanzialmente migliorato l'annotazione del genoma della patata dolce "Taizhong 6", introducendo una versione più completa e dettagliata, v1.0.a2. Questo aggiornamento utilizza 12 librerie di RNA a lunghezza intera Nanopore e 190 librerie di RNA-seq Illumina, con conseguente identificazione di 360 nuovi geni, modifica o aggiunta di 31.771 modelli di geni e nomenclatura genetica perfezionata.
L'annotazione migliorata include profili dettagliati di espressione dei miRNA, a vantaggio degli studi sulla funzionalità genetica della patata dolce e del progresso delle analisi genomiche in tutta la famiglia delle Convolvulaceae, fornendo così una risorsa fondamentale per la futura ricerca agricola e genetica.
La patata dolce (Ipomoea batatas) è una coltura vitale nei paesi in via di sviluppo, poiché fornisce nutrienti essenziali e combatte la carenza di vitamina A in Africa in quanto specie esaploide. La prima sequenza del genoma, la cultivar Taizhong 6, ha utilizzato il sequenziamento Illumina, successivamente migliorato con tecnologie di sequenziamento di terza generazione, portando a un assemblaggio del genoma in scala cromosomica di alta qualità. Tuttavia, permangono delle sfide per ottenere annotazioni precise del genoma a causa delle limitazioni del sequenziamento a lettura breve.
Recentemente, i progressi nel sequenziamento a lunga lettura, come Oxford Nanopore Technologies, hanno facilitato annotazioni più accurate e consentito approfondimenti più approfonditi sulla struttura genetica e sullo splicing alternativo.
Uno studio pubblicato su Piante tropicali il 21 marzo 2024, si basa su questi progressi utilizzando una pipeline di annotazione raffinata che ha incorporato sia set di dati Nanopore a lunghezza intera che estesi di RNA-seq, migliorando l'attuale annotazione del genoma della patata dolce.
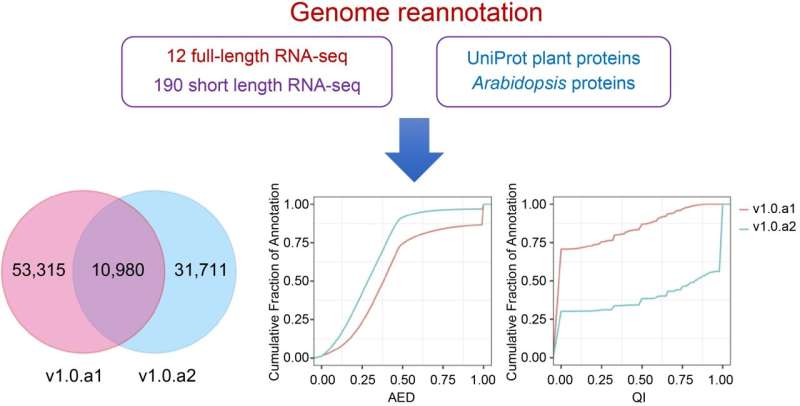
In questo studio, i ricercatori hanno migliorato l'annotazione del genoma di I. batata portandolo alla versione 1.0.a2, utilizzando un approccio completo che integra i trascrittomi a lunghezza intera di Nanopore e i dati RNA-seq Illumina attraverso vari stadi di sviluppo e tessuti della patata dolce.
Il loro metodo ha utilizzato BRAKER per le previsioni genetiche iniziali, arricchite da vari suggerimenti genomici, seguite dalla generazione di modelli di consenso tramite EVidenceModeler (EVM). In particolare, l'annotazione aggiornata ora contiene 42.751 geni codificanti proteine, migliorando il modello con UTR 3' e 5' e aumentando il conteggio medio degli esoni per gene.
Significativamente, questa revisione ha aggiunto o modificato 31.771 modelli genetici, incorporando 8.736 isoforme di splicing alternativo e ha introdotto una nuova nomenclatura genetica per un riferimento più chiaro. Questa annotazione più dettagliata aiuta negli studi genomici precisi e supporta la genomica funzionale avanzata nella patata dolce.
Inoltre, l’integrazione dei dati dei miRNA e dei loro obiettivi offre nuove informazioni sulla regolazione genetica, in particolare durante le diverse fasi di sviluppo delle radici di stoccaggio, migliorando la nostra comprensione della biologia della patata dolce e favorendo gli sforzi di selezione mirati. Le previsioni complete sulla funzione genetica sono state eseguite utilizzando InterProScan e il mappatore EggNOG, fornendo un'annotazione più ricca, fondamentale per la ricerca in corso e i programmi di selezione incentrati sul miglioramento delle cultivar di patata dolce per l'agricoltura globale.
Secondo il ricercatore principale dello studio, il Prof. Guopeng Zhu, "Il nostro studio contribuisce a un'annotazione aggiornata del genoma della patata dolce, che faciliterà in modo significativo gli studi funzionali genetici nella patata dolce e promuoverà le analisi genomiche in tutta la famiglia delle Convolvulaceae."
Nel complesso, questo quadro genomico potenziato facilita una genomica funzionale più profonda nella patata dolce e supporta programmi di selezione avanzati integrando dati dettagliati sui miRNA e previsioni sulla funzione genetica per migliorare i tratti della cultivar.
Ulteriori informazioni: Bei Liang et al, Riannotazione del genoma della patata dolce (Ipomoea batatas (L.) Lam.) utilizzando estesi set di dati RNA-seq basati su Nanopore e Illumina, Piante tropicali (2024). DOI:10.48130/tp-0024-0009
Fornito dall'Accademia cinese delle scienze