L’inizio della vita sulla Terra e la sua evoluzione nel corso di miliardi di anni continuano ad affascinare i ricercatori di tutto il mondo. Il dogma centrale o il flusso direzionale delle informazioni genetiche da un modello di acido desossiribonucleico (DNA) a una trascrizione di acido nucleico ribosio (RNA) e infine in una proteina funzionale, è fondamentale per la struttura e le funzioni cellulari.
Il DNA funziona come il modello della cellula e trasporta le informazioni genetiche necessarie per la sintesi delle proteine funzionali. Al contrario, le proteine sono necessarie per la sintesi del DNA. Pertanto, se sia emerso prima il DNA o le proteine, continua a rimanere una questione di dibattito.
Questa versione molecolare della questione “dell’uovo e della gallina” portò alla proposta di un “Mondo a RNA”. Gli RNA sotto forma di "ribozimi" o enzimi RNA trasportano informazioni genetiche simili al DNA e possiedono anche funzioni catalitiche come le proteine.
La scoperta dei ribozimi alimentò ulteriormente l'ipotesi RNA World secondo cui l'RNA svolgeva la duplice funzione di "immagazzinamento delle informazioni genetiche" e "catalisi", facilitando le attività della vita primitiva esclusivamente tramite RNA. Mentre i ribosomi moderni sono un complesso di RNA e proteine, i ribosomi durante le prime fasi evolutive potrebbero essere stati messi insieme attraverso l'assemblaggio di singole unità funzionali di RNA.
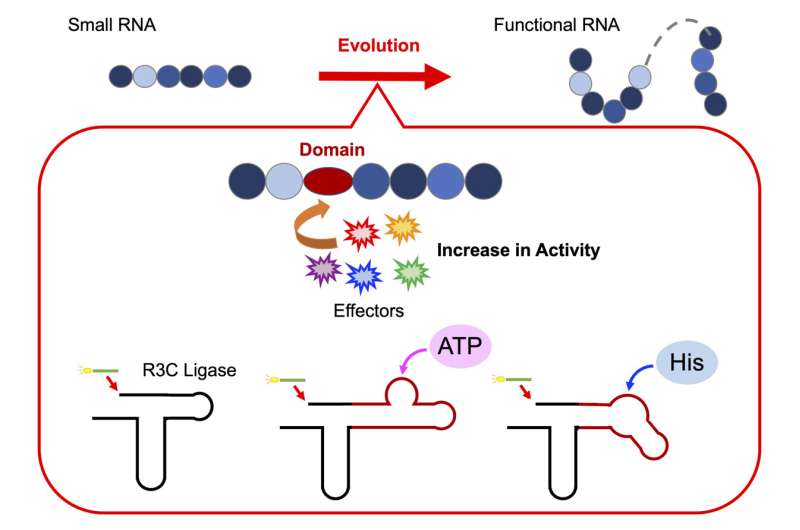
Per verificare questa ipotesi, il professor Koji Tamura, insieme al suo team di ricercatori del Dipartimento di Scienze e Tecnologie Biologiche dell'Università delle Scienze di Tokyo, ha condotto una serie di esperimenti per decodificare l'assemblaggio dei ribozimi funzionali. A tale scopo, hanno progettato un ribozima artificiale, la ligasi R3C, per studiare come le singole unità di RNA si uniscono per formare una struttura funzionale.
Il loro lavoro è pubblicato sulla rivista Life .
Fornendo ulteriori informazioni sulla loro ricerca, il Prof. Tamura afferma:"La ligasi R3C è un ribozima che catalizza la formazione di un legame 3',5'-fosfodiestere tra due molecole di RNA. Abbiamo modificato la struttura aggiungendo domini specifici che possono interagire con vari effettori."
All'interno dei ribosomi, che sono il sito della sintesi proteica, le unità di RNA si assemblano per funzionare come centri della peptidil transferasi (PTC) in modo tale da formare un'impalcatura per il reclutamento degli amminoacidi (singoli componenti di un peptide/proteina) attaccati ai tRNA .
Si tratta di una visione importante della storia evolutiva dei sistemi di sintesi proteica, ma non è sufficiente per tracciare il percorso evolutivo basato sull'ipotesi del mondo dell'RNA.
Per esplorare se l'allungamento dell'RNA, ottenuto collegando insieme le singole unità di RNA, è regolato allostericamente, i ricercatori hanno alterato la struttura della ligasi R3C. Lo hanno fatto incorporando brevi sequenze di RNA che legano l’adenosina trifosfato (ATP), una molecola portatrice di energia vitale nelle cellule, nel ribozima. Il team ha notato che l'attività della ligasi R3C dipendeva dalla concentrazione di ATP, con un'attività maggiore osservata a concentrazioni più elevate di ATP.
Inoltre, un aumento della temperatura di fusione (Tm valore) ha indicato che il legame dell'ATP alla ligasi R3C ha stabilizzato la struttura, il che probabilmente ha influenzato la sua attività di ligasi.
Allo stesso modo, fondendo una sequenza di RNA che lega la L-istidina al ribozima, hanno notato un aumento dell'attività della ligasi all'aumentare delle concentrazioni di istidina (un amminoacido chiave). In particolare, l’aumento dell’attività era specifico all’aumento delle concentrazioni di ATP o istidina; non sono stati osservati cambiamenti in risposta ad altri nucleotidi trifosfati o amminoacidi.
Questi risultati suggeriscono che l'ATP e l'istidina agiscono come molecole effettrici che innescano cambiamenti conformazionali strutturali nel ribozima, che influenzano ulteriormente la stabilità e l'attività dell'enzima.
L'ATP è il vettore energetico centrale della cellula che supporta numerosi processi molecolari, mentre l'istidina è l'amminoacido più comune che si trova nel sito attivo degli enzimi e ne mantiene la chimica acido-base. Considerati gli importanti ruoli dell'ATP e dell'istidina nelle interazioni dell'RNA e nelle funzioni molecolari, questi risultati forniscono nuove informazioni sul ruolo dell'RNA nelle prime fasi dell'evoluzione, inclusa l'origine del codice genetico.
Inoltre, ribozimi ingegnerizzati come quello sviluppato in questo studio sono promettenti in una miriade di applicazioni, tra cui la somministrazione mirata di farmaci, terapie, nanobiosensori, ingegneria enzimatica e sintesi di nuovi enzimi con usi in vari processi industriali.
Nel complesso, questo studio può offrire spunti su come è avvenuta la transizione dal mondo dell'RNA al moderno "mondo del DNA/proteine". Una comprensione fondamentale del mondo dell'RNA, a sua volta, può migliorarne l'uso nelle applicazioni della vita reale.
"Questo studio porterà alla delucidazione del processo di 'acquisizione di funzione e cooperatività basata sull'allostericità' nell'evoluzione dell'RNA. Le interazioni RNA-RNA, le interazioni RNA-amminoacido e l'allostericità applicate in questa ricerca possono guidare la fabbricazione di arbitrari Nanostrutture di RNA, con varie applicazioni", conclude il Prof. Tamura.
Ulteriori informazioni: Yuna Akatsu et al, Sviluppo di ribozimi allosterici per ATP e l-istidina basati sul ribozima della ligasi R3C, Life (2024). DOI:10.3390/vita14040520
Fornito dall'Università delle Scienze di Tokyo