La ricerca congiunta condotta da Yutaro Shuto, Ryoya Nakagawa e Osamu Nureki dell'Università di Tokyo ha determinato la struttura spaziale di vari processi di un nuovo strumento di modifica genetica chiamato "prime editor". L'analisi funzionale basata su queste strutture ha anche rivelato come un "editor primario" potrebbe ottenere la trascrizione inversa, sintetizzando il DNA dall'RNA, senza "tagliare" entrambi i filamenti della doppia elica.
Chiarire questi meccanismi molecolari contribuisce notevolmente alla progettazione di strumenti di editing genetico sufficientemente accurati per i trattamenti di terapia genica. I risultati sono pubblicati sulla rivista Nature .
Il Premio Nobel per la Chimica 2020 è stato assegnato a Jennifer Doudna ed Emmanuelle Charpentier per aver sviluppato un modo innovativo ma semplice per modificare il DNA, il "progetto" degli organismi viventi. Sebbene la loro scoperta abbia aperto nuove strade alla ricerca, l’accuratezza del metodo e le preoccupazioni sulla sicurezza nel “tagliare” entrambi i filamenti del DNA ne hanno limitato l’uso per i trattamenti di terapia genica. Pertanto, sono in corso ricerche per sviluppare strumenti che non presentino questi inconvenienti.
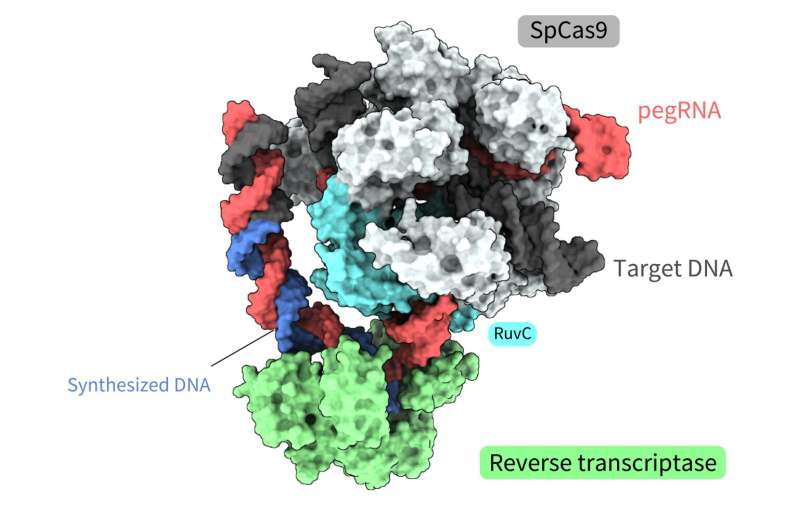
Il sistema di editing principale è uno di questi strumenti, un complesso molecolare costituito da due componenti. Un componente è l'editor principale, che combina una proteina SpCas9, utilizzata nella prima tecnologia di editing genetico CRISPR-Cas, e una trascrittasi inversa, un enzima che trascrive l'RNA in DNA.
Il secondo componente è il prime editing guide RNA (pegRNA), un RNA guida modificato che identifica la sequenza target all'interno del DNA e codifica la modifica desiderata. In questo complesso, l'editor principale funziona come un "elaboratore di testi", sostituendo accuratamente le informazioni genomiche. Lo strumento è già stato implementato con successo in cellule viventi di organismi come piante, pesci zebra e topi. Tuttavia, il modo preciso in cui questo complesso molecolare esegue ogni fase del processo di modifica non è chiaro, principalmente a causa della mancanza di informazioni sulla sua struttura spaziale.
"Siamo diventati curiosi di sapere come funziona insieme la combinazione innaturale di proteine Cas9 e trascrittasi inversa", afferma Shuto, il primo autore dell'articolo.
Il gruppo di ricerca ha utilizzato la microscopia elettronica criogenica, una tecnica di imaging che rende possibili osservazioni su scala quasi atomica. Il metodo richiedeva che i campioni si trovassero in ghiaccio vetroso per proteggerli dal potenziale danno causato dai fasci di elettroni, ponendo alcune sfide aggiuntive.
"Abbiamo riscontrato che il complesso dell'editor principale era instabile in condizioni sperimentali", spiega Shuto. "Quindi è stato molto impegnativo ottimizzare le condizioni affinché il complesso rimanesse stabile. Per molto tempo siamo riusciti solo a determinare la struttura di Cas9."
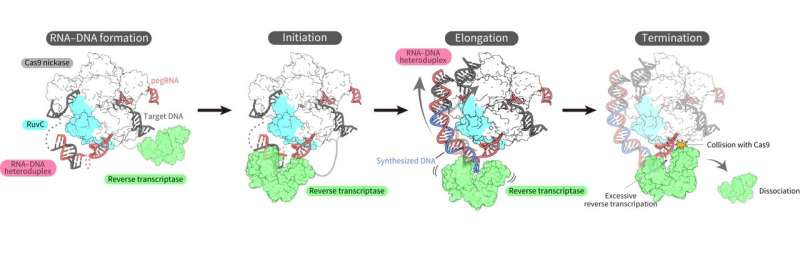
Alla fine, superando le sfide, i ricercatori sono riusciti a determinare la struttura tridimensionale del complesso dell'editor principale in più stati durante la trascrizione inversa sul DNA bersaglio.
Le strutture hanno rivelato che la trascrittasi inversa si lega al complesso RNA-DNA che si forma lungo la "parte" della proteina Cas9 associata alla scissione del DNA, la scissione di un singolo filamento della doppia elica. Durante l'esecuzione della trascrizione inversa, la trascrittasi inversa ha mantenuto la sua posizione rispetto alla proteina Cas9. Le analisi strutturali e biochimiche hanno anche indicato che la trascrittasi inversa potrebbe portare ad ulteriori inserimenti indesiderati.
Questi risultati hanno aperto nuove strade sia per la ricerca di base che per quella applicata. Quindi, Shuto illustra i passaggi successivi.
"La nostra strategia di determinazione della struttura in questo studio può essere applicata anche a redattori principali composti da una diversa proteina Cas9 e trascrittasi inversa. Vogliamo utilizzare le informazioni strutturali appena ottenute per portare allo sviluppo di redattori principali migliorati."
Ulteriori informazioni: Basi strutturali per la trascrizione inversa guidata da pegRNA da parte del principale editore, Nature (2024). DOI:10.1038/s41586-024-07497-8, https://www.nature.com/articles/s41586-024-07497-8
Informazioni sul giornale: Natura
Fornito dall'Università di Tokyo