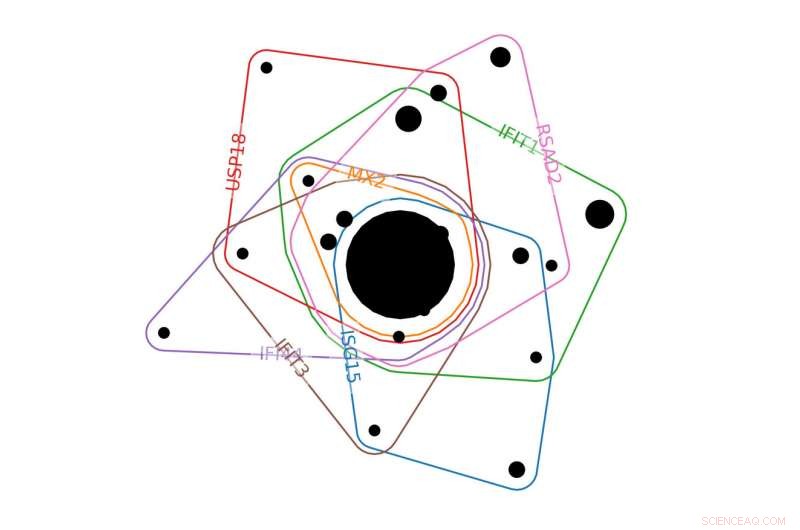
In questa porzione di un esempio di ipergrafo di un sistema biologico, le linee colorate rappresentano i geni e circondano le condizioni sperimentali (cerchi neri) in cui il gene era significativo. Più grande è il cerchio nero, le condizioni più sperimentali sono in quel gruppo. Credito:Emilie Purvine | PNNL
Gli scienziati stanno correndo per tenere il passo con COVID-19, creare nuovi strumenti per capire come funziona il nuovo coronavirus.
Per i ricercatori del Pacific Northwest National Laboratory (PNNL), comprendere l'infezione virale è una questione di matematica piuttosto che un'analisi puramente molecolare. Stanno usando uno strumento matematico avanzato chiamato ipergrafi per identificare come le cellule umane rispondono all'infezione virale, compreso il nuovo coronavirus. Le proteine chiave che partecipano a tale risposta potrebbero essere bersagli per lo sviluppo di farmaci per il trattamento del COVID-19.
La matematica del PNNL Emilie Purvine e il biologo computazionale Jason McDermott hanno recentemente presentato il loro lavoro virtualmente al SIGKDD (Special Interest Group on Knowledge Discovery and Data Mining) dell'Association for Computing Machinery. una conferenza annuale per il data mining, scienza dei dati, e analisi.
Ipergrafi per infezione virale
In un passaggio chiave, il team ha testato il nuovo approccio con i dati di un virus simile, il coronavirus che causa la sindrome respiratoria acuta grave, o SARS. Quel virus ha infettato più di 8, 000 persone come ha spazzato tutto il mondo nel 2003.
Il team del PNNL ha scoperto che i risultati del nuovo metodo corrispondevano ai dati precedentemente raccolti su quel virus. Usando gli ipergrafi, il team ha identificato e classificato l'attività di diversi geni ora noti per essere importanti per l'attività del virus che ha causato l'epidemia di SARS-1.
"Il nostro lavoro ha identificato in modo indipendente gli stessi geni noti per essere importanti con l'attività della SARS. Questo è stato un passo importante da compiere prima di applicare il nostro lavoro al virus che causa il COVID-19, ", ha detto McDermott.
Ora il team PNNL sta applicando la nuova tecnologia al virus attuale, utilizzando ipergrafi per ordinare e classificare l'importanza di molte delle centinaia di geni attivi nel COVID-19.
Purvine e McDermott hanno utilizzato gli ipergrafi per esplorare come le cellule umane rispondono alle infezioni virali negli ultimi due anni. Hanno lavorato con i dati raccolti dalla biologa del PNNL Katrina Waters, che ha monitorato l'espressione genica, espressione proteica, e cambiamenti molecolari nelle cellule umane infettate da virus inclusa l'influenza, Zika, Ebola, e coronavirus per circa un decennio.
Per applicare ipergrafi a questo grande set di dati, i ricercatori hanno prima dovuto capire come identificare i gruppi di proteine in modo da configurarli per costruire un ipergrafo significativo. La squadra stava affrontando quella sfida all'inizio di quest'anno, allo stesso tempo ha colpito la pandemia di coronavirus.
Dai grafici agli ipergrafi
La collaborazione con Purvine offre un nuovo strumento a McDermott, che ha utilizzato tecniche matematiche basate su grafici per analizzare le connessioni tra geni, proteine, e molecole di segnalazione nelle cellule per anni.
Lui ei suoi colleghi identificano le relazioni tra due molecole alla volta. Quindi classificano le connessioni tra molte interazioni separate. Queste connessioni si intrecciano rapidamente in grafici complessi che rappresentano reti molecolari che mantengono in funzione le cellule.
I ricercatori analizzano la struttura e la forma di quei grafici, alla ricerca di modelli significativi che indichino componenti molecolari con ruoli chiave. Centralità, o quando una molecola ha molte connessioni con altre, è un tipo di modello.
L'intera struttura di un grafico è un altro modello significativo. Alcune connessioni centrali fungono da ponti per mantenere il flusso di informazioni tra le diverse parti della rete. I geni o le proteine coinvolte in queste connessioni "intermedie" probabilmente mantengono il corretto funzionamento di un'intera cellula.
Gli ipergrafi rappresentano un potenziale balzo in avanti. Invece di rappresentare le connessioni tra i singoli componenti, gli ipergrafi mostrano le relazioni tra gruppi di cose. Poiché le reti biologiche operano attraverso gruppi molecolari, gli scienziati ritengono che gli ipergrafi potrebbero rappresentare la loro struttura in modo più realistico rispetto ai grafici standard.
Gli scienziati hanno utilizzato ipergrafi per rappresentare gruppi sociali e infrastrutture di rete di computer, ma la loro complessità computazionale li rende una tecnica non comune per lo studio di reti biologiche su larga scala che derivano da dati sperimentali.
Uno strumento software ipergrafo open source chiamato HyperNetX, sviluppato al PNNL, rende questa analisi più accessibile ai ricercatori di varie discipline. Ma l'applicazione della tecnica ai dati provenienti da una varietà di campi richiede ancora qualche ritocco.
"Dato che ci sono tanti modi per costruire ipergrafi da dati biologici, i biologi probabilmente hanno bisogno di coinvolgere un matematico computazionale per farlo, per adesso, " ha detto Purvino.