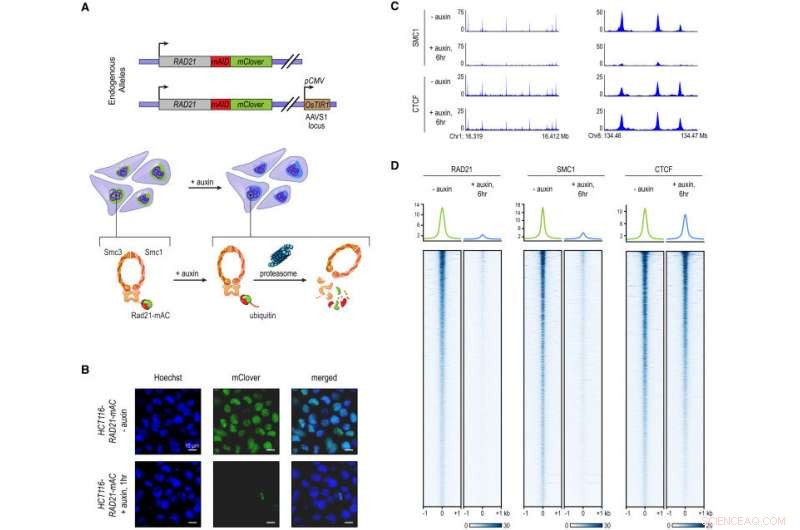
L'etichettatura di RAD21 endogeno con un degron inducibile da auxina consente una rapida, Perdita di coesione quasi completa. (A) Nelle cellule HCT-116-RAD21-mAC, entrambi gli alleli RAD21 sono etichettati con degrons inducibili da auxina e un reporter mClover, e il gene OsTIR1 è integrato nel locus AAVS1. Il trattamento con auxina porta alla degradazione proteasomica di RAD21. (B) Imaging di cellule vive dopo la colorazione Hoechst 33342 per etichettare i nuclei. La fluorescenza nucleare di mClover corrispondente a RAD21 marcata è stata persa dopo 1 ora di trattamento con auxina. (C) Segnale SMC1 e CTCF ChIP-seq con e senza trattamento con auxina. (D) RAD21, SMC1, e segnale CTCF ChIP-seq (sinistra, mezzo, a destra) attraverso tutti i picchi chiamati per ciascuna delle proteine nelle cellule RAD21-mAC non trattate. In alto:Arricchimenti medi per ogni proteina. Dopo la degradazione di RAD21, il complesso della coesione non si lega più alla cromatina. Il legame CTCF non è influenzato. Credito: Cellula . DOI:10.1016/j.cell.2017.09.026
Un team multi-istituzionale che abbraccia il Baylor College of Medicine, Università del riso, La Stanford University e il Broad Institute del MIT e di Harvard hanno creato la prima mappa 4-D ad alta risoluzione del ripiegamento del genoma, tracciare un intero genoma umano mentre si piega nel tempo. Il rapporto, che possono portare a nuovi modi di intendere le malattie genetiche, appare sulla copertina di Cellula .
Fare collegamenti
Per decenni, i ricercatori hanno sospettato che quando una cellula umana risponde a uno stimolo, Gli elementi del DNA che si trovano molto distanti nel genoma si trovano rapidamente l'un l'altro, formazione di anse lungo il cromosoma. Riorganizzando questi elementi del DNA nello spazio, la cellula è in grado di modificare quali geni sono attivi.
Nel 2014, lo stesso team di scienziati ha mostrato che era possibile mappare questi loop. Ma le prime mappe erano statiche, senza la possibilità di guardare i loop cambiare. Non era chiaro se, nello spazio affollato del nucleo, Gli elementi del DNA potrebbero trovarsi abbastanza velocemente da controllare le risposte cellulari.
"Prima, potremmo fare mappe di come il genoma si è piegato quando era in un particolare stato, ma il problema con un'immagine statica è che se non cambia mai nulla, è difficile capire come funzionano le cose, " ha detto Suhas Rao, primo autore del nuovo studio. "Il nostro approccio attuale è più simile alla realizzazione di un film; possiamo guardare le pieghe mentre scompaiono e riappaiono".
Un anello per domarli tutti
Per monitorare il processo di piegatura nel tempo, il team di ricerca ha iniziato interrompendo la coesione, un complesso proteico a forma di anello che si trovava ai confini di quasi tutti i loop conosciuti. Nel 2015, il team ha proposto che la coesione crei anelli di DNA nel nucleo cellulare mediante un processo di estrusione.
"L'estrusione funziona come il regolatore della lunghezza della cinghia su uno zaino, " ha spiegato il dottor Erez Lieberman Aiden, direttore del Center for Genome Architecture presso il Baylor College of Medicine e autore senior del nuovo studio. "Quando fai passare la cinghia da entrambi i lati, forma un anello. Il DNA sembra fare la stessa cosa, tranne per il fatto che gli anelli di coesione sembrano svolgere il ruolo di aggiustatore".
Aiden ha affermato che una previsione cruciale del modello 2015 è che tutti i loop dovrebbero scomparire in assenza di coesione. Nella nuova ricerca, Aiden, Rao e colleghi hanno testato questa ipotesi.
"Abbiamo scoperto che quando abbiamo interrotto la coesione, migliaia di loop sono scomparsi, " disse Rao, uno studente di medicina alla Stanford University, Hertz Fellow e membro del laboratorio di Aiden. "Quindi, quando rimettiamo la coesione, tutti quei loop sono tornati, spesso in pochi minuti. Questo è esattamente ciò che avresti previsto dal modello di estrusione, e suggerisce che la velocità con cui la coesione si muove lungo il DNA è tra le più veloci per qualsiasi proteina umana conosciuta".
Loop contro gruppi
Ma non tutto è andato come si aspettavano i ricercatori. In alcuni casi, loop ha fatto l'esatto contrario di quanto previsto dai ricercatori.
"Mentre abbiamo visto migliaia di anelli del genoma indebolirsi, abbiamo notato uno schema divertente, " disse Aiden, anche uno studioso McNair, Hertz Fellow e ricercatore senior presso il Center for Theoretical Biological Physics della Rice University. "C'erano alcuni loop strani che stavano effettivamente diventando più forti. Poi, mentre rimettiamo la coesione, la maggior parte dei loop si è ripresa completamente, ma questi strani loop hanno fatto di nuovo l'opposto:sono scomparsi!"
Esaminando come le mappe sono cambiate nel tempo, il team si è reso conto che l'estrusione non era l'unico meccanismo che unisce gli elementi del DNA. Un secondo meccanismo, chiamato compartimentazione, non ha coinvolto la coesione.
"Il secondo meccanismo che abbiamo osservato è molto diverso dall'estrusione, " ha spiegato Rao. "L'estrusione tende a riunire gli elementi del DNA due alla volta, e solo se giacciono sullo stesso cromosoma. Questo altro meccanismo può collegare tra loro grandi gruppi di elementi, anche se giacciono su cromosomi diversi. E sembra essere veloce quanto l'estrusione."
Direttore del Broad Institute Eric Lander, un coautore dello studio, disse, "Stiamo iniziando a capire le regole in base alle quali gli elementi del DNA si uniscono nel nucleo. Ora che possiamo tracciare gli elementi mentre si muovono nel tempo, i meccanismi sottostanti stanno iniziando a diventare più chiari".