Le specie animali strettamente imparentate possono sembrare fisicamente diverse, ma potresti essere sorpreso di apprendere che tali differenze possono derivare non solo da cambiamenti nella sequenza del DNA che alterano la struttura o la funzione delle proteine, ma anche perché i cambiamenti nel DNA influenzano il modo in cui tali proteine vengono espresse. In aggiunta a ciò, non tutte le differenze tra le specie possono essere spiegate solo dai cambiamenti nella sequenza del DNA.
La biologa molecolare Emily Hodges, assistente professore di biochimica, studia gli elementi regolatori del nostro genoma ed è interessata ad analizzare il modo in cui i cambiamenti nella sequenza del DNA influenzano la regolazione genetica.
L'espressione dei geni è controllata da elementi regolatori della sequenza del DNA come i potenziatori genici, che aiutano ad aumentare l'espressione di un gene bersaglio.
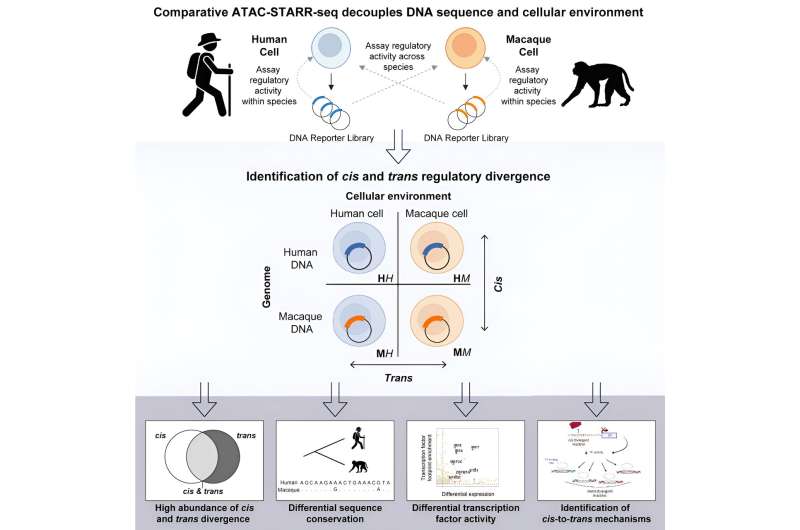
Cambiamenti specie-specifici nella funzione del potenziatore possono derivare da cambiamenti nella sequenza del DNA che si verificano direttamente all'interno di un singolo potenziatore (cis) o all'interno dell'ambiente cellulare in un modo che può influenzare migliaia di potenziatori (trans). Ad esempio, un fattore di trascrizione, una proteina mobile che guida l'espressione di un gene bersaglio, è un elemento transregolatore in grado di legare e controllare potenziatori su diversi cromosomi.
Storicamente, gli scienziati hanno avuto difficoltà a determinare i contributi individuali di questi due meccanismi alla divergenza dell'espressione genetica.
I laboratori di Hodges e dell'ex collega Tony Capra, che ora è professore associato di epidemiologia e biostatistica presso l'Università della California, a San Francisco, hanno utilizzato ATAC-STARR-seq, una tecnica reporter su scala genomica sviluppata dal laboratorio di Hodges, per districare i contributi relativi dei meccanismi di regolamentazione cis e trans alla divergenza di regolamentazione genetica tra gli esseri umani strettamente imparentati e i macachi rhesus. L'articolo "L'evoluzione normativa dei geni umani è guidata dalla divergenza della funzione degli elementi regolatori sia in cis che in trans" è stato pubblicato su Cell Genomics nell'aprile 2024.
Con ATAC-STARR-seq, i ricercatori, guidati dal neolaureato del laboratorio Hodges Tyler Hansen e dalla neolaureata del laboratorio Capra Sarah Fong, hanno esaminato gli effetti di diverse sequenze di DNA (cambiamenti cis) nel contesto di diversi ambienti cellulari (cambiamenti trans) e viceversa e hanno trovato un numero sostanzialmente più elevato di cambiamenti trans nell'attività di regolazione genetica rispetto a quanto osservato in precedenza.
Le differenze tra le specie sono spesso attribuite alla variazione della sequenza (cis), ma i laboratori Hodges e Capra hanno scoperto un ruolo sostanziale per le differenze dell'ambiente cellulare (trans) nel determinare la divergenza di regolamentazione dei geni tra le specie. Questo lavoro sfida l'attuale pensiero secondo cui i cambiamenti regolatori cis sono alla base della maggior parte delle divergenze nell'attività normativa e sostiene un ruolo fondamentale per i cambiamenti regolatori trans nel guidare l'evoluzione regolatoria dei geni.
L'analisi dei contributi relativi dei meccanismi cis e trans della divergenza nella regolamentazione dei geni ha implicazioni per i campi della regolazione dei geni, della genetica delle popolazioni umane e dell'evoluzione dei primati.
Andando avanti, Hodges sta cercando di estendere le scoperte oltre l’evoluzione umana per capire come i meccanismi cis e trans di regolazione genetica contribuiscono alle differenze nel rischio di malattie umane. Queste domande sono fondamentali per comprendere malattie come il cancro, in cui l'interazione tra cambiamenti di sequenza, epigenetica e ambiente cellulare ha un forte impatto sugli esiti della malattia.
Un articolo in anteprima che discute di questa ricerca è stato pubblicato nello stesso numero di Cell Genomics .
Ulteriori informazioni: Tyler J. Hansen et al, L'evoluzione normativa dei geni umani è guidata dalla divergenza della funzione degli elementi regolatori sia in cis che in trans, Cell Genomics (2024). DOI:10.1016/j.xgen.2024.100536
Megan Y. Dennis, Trasformare la nostra comprensione della regolazione genetica specie-specifica, Cell Genomics (2024). DOI:10.1016/j.xgen.2024.100540
Informazioni sul giornale: Genomica cellulare
Fornito dalla Vanderbilt University