Un team combinato di ricercatori medici e specialisti di sistemi di intelligenza artificiale del progetto Deep Mind di Google e Isomorphic Labs, entrambi a Londra, hanno apportato quelli che il gruppo descrive come miglioramenti sostanziali ad AlphaFold 2 che consentono all'applicazione di prevedere la struttura di un'ampia varietà dei sistemi biomolecolari in modo più ampio e accurato. La nuova iterazione si chiama AlphaFold 3.
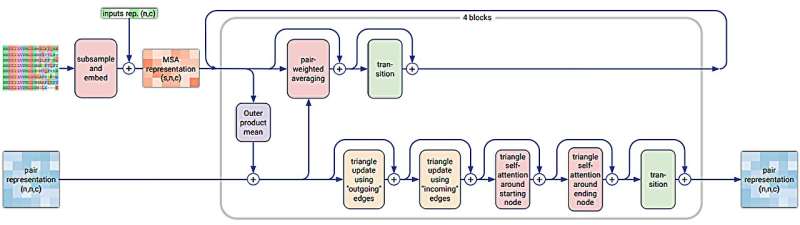
Nel loro studio pubblicato sulla rivista Nature , il gruppo ha utilizzato tecniche di diffusione per apportare miglioramenti al modello architettonico sottostante dell'applicazione per consentire di effettuare previsioni più generali.
La prima versione del sistema di intelligenza artificiale AlphaFold basato sul deep learning è stata rilasciata solo quattro anni fa ed è stata acclamata per la sua capacità di fare previsioni accurate sulla struttura delle proteine utilizzando sequenze di aminoacidi. Ha anche aiutato i ricercatori a comprendere meglio come funzionano le proteine. AlphaFold 2 si è basato su tali capacità, ampliando i complessi che potevano essere previsti.
In questa nuova iterazione, il gruppo di ricerca ha dato all'applicazione la capacità di prevedere sistemi biomolecolari oltre alle proteine. Può prevedere ligandi, ad esempio, o strutture di RNA o DNA. Notano che può anche fare previsioni sulla struttura di ioni, acidi nucleici, altre proteine e sulle interazioni tra anticorpi e antigeni.
Queste capacità, notano i ricercatori, lo rendono uno strumento utile per la scoperta di nuovi farmaci. Una società di scoperta di farmaci (e spin-off di DeepMind) sta già utilizzando il nuovo sistema per fare proprio questo.
Oltre a fare previsioni su altre strutture biomolecolari, il gruppo di ricerca afferma che AlphaFold 3 è anche molto più accurato delle sue iterazioni precedenti e dei suoi concorrenti. Ma riconoscono anche che c’è spazio per crescere:AlphaFold 3 ha un tasso di errore di chiralità del 4,4%, per esempio. A volte ha anche allucinazioni, il che riduce l'aspetto dei nastri.
Notano che il lavoro continuerà con il sistema AlphaFold mentre il team cerca di migliorare la precisione e aggiungere più tipi di sistemi a cui può essere applicato. Hanno inoltre in programma di introdurre una struttura di classificazione per aiutare gli utenti a esprimere giudizi sui risultati forniti dal sistema.
Ulteriori informazioni: Josh Abramson et al, Previsione accurata della struttura delle interazioni biomolecolari con AlphaFold 3, Nature (2024). DOI:10.1038/s41586-024-07487-w
Informazioni sul giornale: Natura
© 2024 Rete Scienza X